Endonucleasa homing

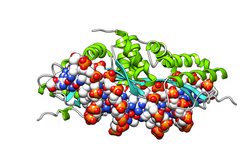
Homodímero de I-CreI sobre el DNA: la enzima se muestra en todo su volumen, cada monómero de un color, rosa o amarillo. Los átomos del DNA se muestran en esferas, coloreadas según cada elemento químico.
| Endonucleasa homing | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | ||||
| Identificadores | ||||
| Identificadores externos |
Bases de datos de enzimas
| |||
| Número EC | 3.2 | |||
| Estructura/Función proteica | ||||
| Tipo de proteína | Hidrolasa | |||
| Funciones | Enzima | |||
| Ortólogos | ||||
| Especies |
| |||
| Ubicación (UCSC) |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
Las endonucleasas homing o 'buscador de objetivos de endonucleas'a (adaptación al español del término inglés homing endonucleases)[1] son un tipo especial de enzimas de restricción codificadas por intrones o inteínas, que actúan sobre el DNA de la propia célula que las sintetiza. Más concretamente, en el alelo opuesto al gen que las codifica. La palabra homing alude al movimiento o traslado de esos intrones o inteínas hacia dicho alelo.[2]
Con respecto a las enzimas de restricción tradicionales (en adelante "enzimas de restricción"), las endonucleasas homing se diferencian en:[3]
- Unirse a secuencias de reconocimiento asimétricas y de gran longitud (de 12 a 40 pb), mientras que las enzimas de restricción se unen a secuencias de reconocimiento cortas (de 3 a 8 pb), normalmente simétricas.
- Toleran cierta degeneración en su secuencia de reconocimiento, es decir, ligeras variaciones en ésta disminuyen la actividad de la enzima, pero no la inhiben completamente.[4][5]
- Estructuralmente comparten motivos que permiten agruparlas en 4 familias diferentes, mientras que en las enzimas de restricción clásicas, por su lado, no se han determinado familias claramente reconocibles.
- La naturaleza de sus asociaciones moleculares: estas enzimas actúan como monómeros u homodímeros, a menudo unidas a otras proteínas asociadas, o bien a moléculas de RNA formando ribonucleoproteínas.[6] Mientras que las enzimas de restricción, cuando necesitan de subunidades accesorias, requieren otro tipo de cofactores.
- Su distribución filogenética: estas enzimas han sido encontradas en los tres dominios de seres vivos (arqueas, bacterias y eucariotas) mientras que las enzimas de restricción clásicas sólo en células procariontes (bacterias y arqueas) y algunos tipos de virus eucariotas.[7][8]
Nomenclatura
[editar]- Para una lista exhaustiva, consultar el artículo principal Anexo:Endonucleasas homing de restricción.
El sistema de nomenclatura que se ha adoptado es similar al de las enzimas de restricción con algunos añadidos:
- Se indica en primer lugar un prefijo seguido de guion, "I-" si la enzima es codificada por un intrón o "PI-" si lo es por una inteína (del inglés "protein insert", literalmente "inserto de proteína").
- A continuación, tres letras indican el género y la especie del organismo que la produce, de las cuales, la primera en mayúsculas corresponde al género y las dos siguientes, en minúsculas, a la especie.
- Y finalmente un número romano distingue las múltiples enzimas encontradas dentro del mismo organismo.
Como ejemplo, podemos citar la enzima PI-TliII, segunda enzima codificada en una inteína descubierta en el organismo Thermococcus litoralis; o H-DreI, primera endonucleasa homing artificial creada en el laboratorio a partir de las enzimas I-DmoI y I-CreI de Desulfurococcus mobilis y Chlamydomonas reinhardtii, respectivamente.
Origen y mecanismo de funcionamiento
[editar]Aunque el origen y la función de las endonucleasas homing todavía se encuentra en proceso de investigación, las últimas hipótesis los consideran como elementos genéticos egoístas,[10] tales como los transposones, ya que estas enzimas facilitan la autoperpetuación del elemento genético al que pertenecen sin que haya sido muy claro determinar que esto ofrece una ventaja selectiva al organismo:
La secuencia de reconocimiento a la que se unen es lo suficientemente larga como para que su presencia aparezca al azar con una probabilidad muy reducida (aproximadamente cada 7•1010 pb),[11] encontrándose habitualmente una única repetición de esta secuencia por genoma, concretamente en el gen que codifica la endonucleasa (HEG, de "homing endonuclease gene"). Este HEG se encuentra precisamente en medio de la secuencia de reconocimiento de la enzima que codifica, interrumpiéndola e impidiendo por tanto el corte en el DNA que lo porta.
En la situación inicial previa a la transmisión, un alelo porta el gen (el cual es denominado "alelo HEG+") mientras que el otro no ("alelo HEG-"), siendo por tanto susceptible este último de ser cortado por la enzima. Una vez la enzima es sintetizada ejerce su acción rompiendo el cromosoma en el alelo HEG-, a lo cual responde el sistema de reparación del DNA celular por medio de recombinación, utilizando como patrón el DNA intacto del alelo opuesto HEG+ para reparar el daño. Este DNA que es utilizado como patrón contiene el gen de la endonucleasa. De esta manera, el gen es copiado en el alelo que inicialmente no lo tenía.[12]
Este proceso es conocido por su denominación anglosajona "homing".[12]
Familias estructurales
[editar]
| ||||||||||||||||
Actualmente, se reconocen 4 diferentes familias estructurales, atendiendo a sus motivos conservados:[3]
- LAGLIDADG: Contienen 1 o 2 motivos LAGLIDADG. La secuencia LAGLIDADG es una secuencia conservada de aminoácidos donde cada letra corresponde a un residuo. Esta secuencia interviene directamente en la catálisis de la actividad enzimática, es decir, en el corte del DNA. Aquellas enzimas que poseen un único motivo, forman homodímeros creando una hendidura en su interfase que interacciona con el surco menor del DNA. En esta hendidura se disponen los dos motivos LAGLIDADG. Las enzimas que poseen dos motivos, actúan como monómeros creando una hendidura de manera similar.
- GIY-YIG: Contienen un único motivo CIY-YIG, en la región N-terminal, que interacciona con el DNA en el sitio de corte. La enzima prototípica de esta familia, I-TevI, actúa como monómero.
- His-Cys box: Las enzimas de esta familia contienen una región de 30 aminoácidos en la que se incluyen 5 aminoácidos conservados: dos histidinas y tres cisteínas; que coordinan el catión metálico necesario para la catálisis. I-PpoI es la enzima mejor caracterizada de esta familia, y actúa como monómero.
- H-N-H: Contienen una secuencia consenso de aproximadamente 30 aminoácidos, que incluye dos parejas de histidinas conservadas y una asparragina, que forman un domino "dedo de zinc".
Véase también
[editar]- Enzima de restricción.
- Anexo:Endonucleasas homing de restricción.
- Anexo:Lista de sitios de corte de enzimas de restricción.
- Intrones e inteínas.
- Transposón.
- Genética dirigida
Enlaces externos
[editar]- Base de datos exhaustiva de endonucleasas, hospedada en la página web de New England Biolabs©. Incluye toda clase de información biológica, estructural, cinética y comercial acerca de miles de enzimas. También incluye referencias bibliográficas específicas para cada molécula: Roberts RJ, Vincze T, Posfai, J, Macelis D. «REBASE» (en inglés). Consultado el 3 de febrero de 2010. «Restriction Enzyme Database».
- Base de datos de inteínas, alojada en la página web de New England Biolabs©. Perler FB. «InBase» (en inglés). Archivado desde el original el 2 de agosto de 2010. Consultado el 5 de febrero de 2010. «The Intein Database and Registry»..[14]
Referencias
[editar]- ↑ Cf. Nodo Nacional de Bioinformática, «Enzimas y nomenclatura enzimática» (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última)., consultado el 13-9-2010.
- ↑ Cf. Barry L. Stoddard, «Homing endonuclease structure and function», Quarterly Reviews of Biophysics, 38, 1 (2006), págs. 49–95 (50).
- ↑ a b c Belfort M and Roberts RJ (septiembre de 1995). «Homing endonucleases: keeping the house in order». Nucleic Acids Res 25 (17): 3379-88. PMID 9254693. doi:10.1093/nar/25.17.3379.
- ↑ Gimble FS, Wang J (octubre de 1996). «Substrate recognition and induced DNA distortion by the PI-SceI endonuclease, an enzyme generated by protein splicing». J Mol Biol 263 (2): 163-80. PMID 8913299. doi:10.1006/jmbi.1996.0567.
- ↑ Argast GM, Stephens KM, Emond MJ, Monnat RJ Jr (julio de 1998). «I-PpoI and I-CreI homing site sequence degeneracy determined by random mutagenesis and sequential in vitro enrichment». J Mol Biol 280 (3): 345-53. PMID 9665841. doi:10.1006/jmbi.1998.1886.
- ↑ Zimmerly S, Guo H, Eskes R, Yang J, Perlman PS, Lambowitz AM (noviembre de 1995). «A group II intron RNA is a catalytic component of a DNA endonuclease involved in intron mobility». Cell 83 (4): 529-38. PMID 7585955. doi:10.1016/0092-8674(95)90092-6.
- ↑ Roberts RJ, Macelis D (enero de 1997). «REBASE-restriction enzymes and methylases». Nucleic Acids Res 25 (1): 248-62. PMID 9016548.
- ↑ Lambowitz AM, Belfort M (1993). «Introns as mobile genetic elements». Annu Rev Biochem 62: 587-622. PMID 8352597. doi:10.1146/annurev.bi.62.070193.003103.
- ↑ Chevalier BS, Kortemme T, Chadsey MS, Baker D, Monnat RJ, Stoddard BL (octubre de 2002). «Design, activity, and structure of a highly specific artificial endonuclease». Moll Cell 10 (4): 895-905. PMID 12419232. doi:10.1016/S1097-2765(02)00690-1.
- ↑ Edgell DR (febrero de 2009). «Selfish DNA: homing endonucleases find a home». Curr Biol 19 (3): R115-R117. PMID 19211047. doi:10.1016/j.cub.2008.12.019.
- ↑ Jasin M (junio de 1996). «Genetic manipulation of genomes with rare-cutting endonucleases». Trends Genet 12 (6): 224-8. PMID 8928227. doi:10.1016/0168-9525(96)10019-6.
- ↑ a b Burt A, Koufopanou V (diciembre de 2004). «Homing endonuclease genes: the rise and fall and rise again of a selfish element». Curr Opin Genet Dev 14 (6): 609-15. PMID 15531154. doi:10.1016/j.gde.2004.09.010.
- ↑ Jurica MS, Monnat RJ, Stoddard BL (octubre de 1998). «DNA recognition and cleavage by the LAGLIDADG homing endonuclease I-CreI». Moll Cell 2 (4): 469-76. PMID 9809068. doi:10.1016/0092-8674(95)90092-6.
- ↑ Perler FB (enero de 2002). «InBase: the Intein Database». Nucleic Acids Res 30 (1): 383-4. PMID 11752343.