Operón lac

El operón lac es un operón requerido para el transporte y metabolismo de la lactosa en la bacteria Escherichia coli, así como en algunas otras bacterias entéricas. Presenta tres genes estructurales adyacentes, un promotor, un regulador y un operador. El operón lac es regulado por varios factores, incluyendo la disponibilidad de glucosa y de lactosa. La regulación génica del operón lac fue el primer mecanismo regulatorio de la expresión genética en ser elucidado, y es utilizado a menudo como un ejemplo clásico de la regulación génica en procariotas.
Antecedentes
[editar]Hasta el año 1960, se pensaba que todos los moldes necesarios para la síntesis proteica se encontraban en el RNA ribosómico (rRNA). Sin embargo, los pesos moleculares de las proteínas varían hasta en dos órdenes de magnitud respecto de los pesos de los rRNA (5S, 16S, y 23S). Este hecho fue puesto de manifiesto por François Jacob y Jacques L. Monod, quienes ya predijeron la existencia del ARN mensajero (mRNA), el cual se sintetizaría a partir de un ADN molde.
Jacob y Monod decidieron estudiar el metabolismo de la lactosa en E. coli, de la cual se sabía que estaba controlada por tres enzimas cuyos genes se encuentran adyacentes en el cromosoma bacteriano.
Una de estas enzimas era la β-galactosidasa, que hidroliza la lactosa en glucosa y galactosa.También, intervienen otras dos enzimas la galactósido permeasa y la tiogalactósido transacetilasa.[1]
Cuando cultivaban bacterias en glucosa como fuente de carbono podían observar cómo la concentración de β-galactosidasa era muy baja. Al sustituir glucosa por lactosa se observaba un rápido aumento en los niveles de β-galactosidasa y de las otras dos enzimas. Al retirar de nuevo la lactosa, los niveles de las tres enzimas disminuían rápida y drásticamente. Este hecho sugería que los moldes para la síntesis de estas enzimas eran muy inestables, sintetizándose y degradándose rápidamente, lo cual no encajaba con la elevada estabilidad de los rRNAs. Tampoco cabía la posibilidad de que el molde fuera de ADN, que también es muy estable.
Tras analizar numerosos mutantes de E. coli que presentaban un control defectuoso en la inducción de estas enzimas (inducción en ausencia de lactosa, no inducción en presencia de lactosa, etc.) propusieron la existencia de un represor que, uniéndose a una secuencia operadora específica del ADN, regularía la concentración de mRNA.
Todos estos estudios, junto a los realizados por André Lwoff con el bacteriófago λ, permitieron a Jacob y Monod proponer en 1961 una hipótesis unificadora para la regulación génica, llamada posteriormente modelo del operón. Dicho modelo proponía la existencia de represores capaces de unirse a operadores en la secuencia del ADN, los cuales controlaban así la síntesis del mRNA. El mRNA sería una copia complementaria de la secuencia del ADN que codificaba una o varias proteínas (según si hubiera uno o más genes). Al conjunto de genes contiguos más los elementos reguladores que controlan su expresión se le denominó operón.
Estructura y elementos del operón lac
[editar]El operón lactosa presenta los siguientes elementos:[2]
- Genes estructurales:
- Gen lac z: codifica la enzima β-galactosidasa, que cataliza la reacción de hidrólisis de la lactosa en glucosa y galactosa.
- Gen lac y: codifica la proteína galactósido permeasa, cuya función es facilitar el transporte de la lactosa al interior de la bacteria colocándose en la membrana plasmática y formando un carrier (proteína transportadora).
- Gen lac a: codifica la enzima tiogalactósido transferasa, que cataliza la transferencia del grupo acetil del acetil Coenzima A al 6-OH de un aceptor tiogalactósido. Este gen no está relacionado con el metabolismo de la lactosa.
- Promotor: región del DNA, precedente a los genes estructurales, que reconoce la RNA polimerasa para llevar a cabo la transcripción.
- Operador: región del DNA localizada entre el promotor y el comienzo de los genes estructurales, que es reconocida por la proteína represora Lac I.
- Gen represor (lac I): codifica la proteína represora Lac I, que reconoce la región operadora, donde se une. Impide la transcripción de los genes bajo el control de este promotor pero estimula la unión de la RNA polimerasa formando lo que se conoce como Complejos Cerrados. Cuando el represor se retire (en presencia de inductor que en este caso será lactosa o IPTG), la RNA polimerasa estará lista para formar Complejos Abiertos y empezar la transcripción.

Regulación del operón lac
[editar]El operón lac se encuentra bajo un tipo de regulación negativa, donde los genes pueden transcribirse siempre, salvo cuando la proteína represora Lac I se encuentra unida a la región operadora, por la cual presenta una elevada afinidad. En este caso, el promotor del gen lac I es constitutivo, por lo que la proteína Lac I se expresa permanentemente y permanece unida en forma de tetrámero a la zona operadora, impidiendo la transcripción de los genes estructurales. La regulación del operón funcionaría de la siguiente forma:[3]
- En presencia de lactosa: la lactosa es el inductor del operón. Es capaz de unirse a la proteína represora Lac I y generar un cambio conformacional que disminuye su afinidad por la región operadora. De esta forma, la región operadora queda libre, la RNA polimerasa puede transcribir libremente los genes estructurales y la β-galactosidasa puede degradar la lactosa a glucosa más galactosa.
- En ausencia de lactosa: en ausencia de inductor, la proteína represora Lac I mantiene su elevada afinidad por la región operadora, impidiendo que la RNA polimerasa transcriba los genes estructurales. De esta forma, el sistema permanece cerrado con el consecuente ahorro energético para la bacteria.

La verdadera molécula inductora del operón lac es la alolactosa, un isómero de la lactosa que se obtiene por una transglicosilación llevada a cabo ocasionalmente por la β-galactosidasa.
El mRNA de los genes estructurales presenta una vida media de unos tres minutos, lo que permite que la inducción pueda revertirse rápidamente en ausencia de lactosa.
Por último, cabe destacar que el operón lac presenta una expresión basal, al igual que la mayoría de los operones. Esto quiere decir que, en ausencia de inductor, el operón puede expresar los genes estructurales en muy baja concentración y así mantener unas bajas concentraciones de β-galactosidasa y de permeasa. La permeasa permitirá que pueda entrar inicialmente la lactosa en la bacteria y la β-galactosidasa permitirá transglicosilar la lactosa y transformarla en alolactosa, el verdadero inductor del operón lac. A medida que entre más lactosa en la bacteria, se irán expresando en mayor medida los genes estructurales.
Represión catabólica
[editar]El operón lac presenta además otro tipo de regulación que viene mediada por el AMP cíclico y la proteína CRP (catabolite repressor protein). A lo largo de todo su estudio, Jacob y Monod también pudieron observar cómo las bacterias eran capaces de seleccionar la fuente de carbono que utilizaban cuando tenían más de una a su alcance.[4] Este comportamiento era lógico desde el punto de vista del ahorro energético, pero ¿cómo hacían las bacterias para «elegir»?
Cuando las bacterias crecen en glucosa más lactosa como fuentes de carbono, primero utilizan la glucosa y solo después, tras haber agotado la glucosa, utilizan la lactosa, en lo que se denomina crecimiento diaúxico. Además, se puede observar que, a pesar de estar el inductor (lactosa) presente, no hay transcripción de los genes estructurales del operón lac.
La respuesta se encontró al observar que la transcripción de los genes estructurales no dependía únicamente de la ausencia del represor Lac I en la región operadora, sino que además era necesario que se uniera la proteína CRP más AMPc alrededor de la región -60, respecto del comienzo del gen lac Z. La proteína CRP se encuentra en forma de dímero y solo es activa cuando lleva unido AMPc. El alto contenido de AMPc se debe a que en ausencia de transporte de glucosa al interior celular se aumenta la actividad de la adenilato ciclasa (enzima encargada de la síntesis de AMPc). Aquellos mutantes para la adenilato ciclasa (que no producen AMPc) o para la proteína CRP presentaban niveles muy bajos de expresión de los genes del operón lac. Por lo tanto, sin CRP o sin AMPc el sistema permanecería cerrado. Esto unido al hecho de que la glucosa disminuía los niveles de AMPc daba la explicación por la cual la glucosa ejercía una represión por catabolito. Este tipo de represión se ha podido observar también en el operón de la arabinosa, de la galactosa, de la maltosa... De este modo, la presencia de glucosa inhibe de forma conjunta muchos operones que regulan la utilización de fuentes alternativas de carbono y energía, es decir, la represión catabólica constituye un regulón.
Análogos de la lactosa
[editar]Se han encontrado una serie de moléculas derivadas o análogas de la lactosa, las cuales han sido de gran utilidad a la hora de trabajar con el operón lac en el laboratorio. La mayoría de estos compuestos son galactósidos derivados de la lactosa, donde la glucosa ha sido sustituida por algún radical o grupo químico. A continuación se pueden observar los análogos de lactosa más representativos:
- IPTG (isopropil-β-D-tiogalactósido): suele utilizarse como inductor artificial del operón lac, ya que es capaz de unirse al represor LacI, pero no es un sustrato para la β-galactosidasa y no puede ser metabolizado por la bacteria. Además, el IPTG es transportado eficientemente al interior de la bacteria en ausencia de la permeasa, con lo que su entrada es independiente de la expresión del gen lac y.
- Fenil-Gal (fenil-β-D-galactosa): es un sustrato de la β-galactosidasa, pero no es un inductor del operón, ya que es incapaz de unirse al represor LacI. Esto hace que las cepas bacterianas silvestres sean incapaces de crecer cuando su única fuente de carbono es fenil-gal. Solo aquellos mutantes donde se encuentre ausente el represor LacI podrán crecer en esta fuente de carbono.
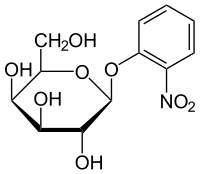
- ONPG (orto-nitrofenil-β-D-galactopiranósido): es un sustrato de la β-galactosidasa que, al ser hidrolizado, produce un compuesto (ortonitrofenol) que presenta un intenso color amarillo. El ONPG es muy utilizado en los ensayos in vitro de β-galactosidasa, en los que se puede obtener la concentración de β-galactosidasa en función de la intensidad del color amarillo, medida por absorbancia a una longitud de onda de 420 nm.
- X-gal (5-bromo-4-cloro-3-indolil-β-D-galactósido): es otro sustrato de la β-galactosidasa que, al ser hidrolizado, produce un compuesto (indoxil) que en contacto con el aire se transforma en índigo insoluble, el cual presenta un intenso color azul. Es utilizado como indicador de expresión de la β-galactosidasa en colonias bacterianas creciendo en placa. Aquellas colonias que estén expresando la enzima se tornarán de un color azul más o menos intenso en función de la cantidad de enzima que estén expresando.
- Alolactosa: es un isómero de la lactosa y es el verdadero inductor del operón lac. Mientras que la lactosa es galactosa-(β1-4)-glucosa, la alolactosa es galactosa-(β1-6)-glucosa. La lactosa, una vez en el citoplasma bacteriano, es transformada en alolactosa por la β-galactosidasa.
Nomenclatura genética
[editar]Se utilizan códigos de tres letras para describir los fenotipos de las bacterias. Ejemplos de esto son: Lac (capacidad de utilizar lactosa como fuente de carbono), His (capacidad de sintetizar el aminoácido histidina), Mot (motilidad natatoria) y Str (resistencia al antibiótico estreptomicina). En el caso de Lac, las cepas silvestres son Lac+, presentando la capacidad de utilizar lactosa como una fuente de carbono y de energía, y los mutantes Lac-, derivados de ellas que han perdido dicha capacidad y no pueden usar la lactosa.
Generalmente, estas tres letras también son utilizadas (en minúsculas e itálicas) para señalar los genes involucrados en un fenotipo particular, donde cada gen es distinguido por una letra adicional. Los genes lac que codifican para enzimas son lac z, lac y y lac a. El cuarto gen es lac I, que codifica para el represor Lac I del operón lac. Es posible distinguir entre genes estructurales que codifican para enzimas, y genes reguladores que codifican para proteínas que regulan la expresión génica.
El uso actual amplía la nomenclatura fenotípica para aplicarse a proteínas: así, LacZ es el producto proteíco del gen lac z, es decir, la β-galactosidasa. Varias secuencias cortas que no son genes también afectan la expresión génica, incluyendo al promotor lac, lac p, y el operador lac, lac o. A pesar de no ser del todo riguroso, las cepas cuyo lac o se encuentra mutado son denominadas lac oc, por razones históricas.
Clasificación de mutantes regulatorios
[editar]Uno de los avances conceptuales de Jacob y Monod fue el de reconocer la distinción entre sustancias regulatorios y los sitios donde ellas actúan para cambiar la expresión génica. Jacob, un exsoldado, utilizó la analogía de un bombardero que liberaría su carga letal al recibir una transmisión especial de radio, o una señal. Un sistema funcional requiere tanto de un transmisor en tierra como de un receptor en el aeroplano. Ahora bien, supóngase que el transmisor que habitualmente se usa está roto. Podría hacerse funcionar este sistema introduciendo un segundo transmisor, funcional. En contraste, dijo Jacob, considere un bombardero con un receptor defectuoso. El comportamiento de este bombardero no puede ser cambiado por introducción de un segundo aeroplano, funcional.
Para analizar los mutantes regulatorios del operón lac, Jacob desarrolló un sistema en el cual una segunda copia de los genes lac (lacI con supromotor, y lacZYA con promotor y operador) podían ser introducidos en una única célula. Se ensaya entonces el fenotipo regulatorio de un cultivo de tales bacterias, las cuales son diploides para los genes lac, pero en cualquier otro sentido normales. En particular, se determina si LacZ y LacY se producen incluso en la ausencia de IPTG. El experimento, en el cual tanto genes como grupos de genes se testean de a pares, es denominado un test de complementación.

Este test se encuentra ilustrado en la figura (se omite lacA por razones de simplicidad). En principio, se muestran ciertos estados haploides (es decir, la célula solo tiene una única copia de los genes lac). El panel (a) muestra represión, (b) muestra inducción por IPTG, y (c) y (d) muestran el efecto de una mutación al gen lacI o al operador, respectivamente. En el panel (e) se muestra el test de complementación para el represor. Si una copia de los genes lac lleva una mutación en lacI, pero la segunda copia es del tipo salvaje para lacI, el fenotipo resultante es normal —no se expresa LacZ sin IPTG—. Se dice que las mutaciones que afectan al represor son recesivas para el tipo salvaje (y que el tipo salvaje es dominante), y esto se explica por el hecho de que el represor es una pequeña proteína que puede difundir hacia el interior de la célula. La copia del operón lac adyacente al gen lacI defectuoso es apagado efectivamente por la proteína producida por la segunda copia de lacI.
Si el mismo experimento se lleva a cabo utilizando una mutación en el operador, se obtiene un resultado diferente (panel (f)). El fenotipo de una célula que lleva un sitio de operador mutante y uno de tipo salvaje es que LacZ y LacY se producen incluso en la ausencia del inductor IPTG. La mutación del operador es dominante. Cuando el sitio del operador donde el represor debe unirse está dañado por mutación, la presencia de un segundo sitio funcional en la misma célula no hace diferencia alguna a la expresión génica controlada por el sitio mutante.
Una versión más sofisticada de este experimento utiliza operones marcados para distinguir entre las dos copias de los genes lac y mostrar que el/los genes no regulados son quienes se encuentran junto al operador mutante (panel (g)). Por ejemplo, supóngase que se marca una copia con una mutación que inactiva lacZ para que entonces solo se produzca la proteína LacY, mientras que la segunda copia trae una mutación que afecta lacY y solo puede producir LacZ. En esta versión, solo la copia del operón lac adyacente al operador mutante se expresa sin IPTG. Decimos que la mutación del operador es cis-dominante, si es dominante para tipos salvajes pero afecta solo la copia del operón inmediatamente adyacente a ella.
Esta explicación puede ser engañosa en un sentido importante, porque se deriva de una descripción del experimento y luego explica los resultados en términos de un modelo. Pero de hecho, es a menudo cierto que el modelo es planteado en primer lugar, y que luego se diseña un experimento específicamente para comprobar la validez del mismo. Jacob y Monod imaginaron primero que debe haber un sitio en el ADN con las propiedades del operador, y luego diseñaron ensayos de complementación para mostrar esto.
La dominancia de los mutantes del operador sugiere también un procedimiento para selecccionarlos específicamente. Si se selecciona a los mutantes regulatorio de un cultivo de tipo salvaje utilizando fenil-Gal, como se describe anteriormente, las mutaciones del operador son raras comparadas con los mutantes del represor, debido a que el tamaño objetivo es demasiado pequeño. Pero si, en vez de comenzar con una cepa que lleva dos copias de los genes lac (es decir, que sea diploide para lac), las mutaciones del represor (que aún continuarán ocurriendo) no serán recobradas, ya que la complementación por los genes de tipo salvaje confiere un genotipo salvaje, y por lo tanto dominarán las mutaciones de operador.
Regulación por AMP cíclico
[editar]El microorganismo experimental utilizado por François Jacob y Jacques Monod fue la bacteria común de laboratorio, E. coli, pero muchos de los conceptos regulatorios básicos que fueron descubiertos por Jacob y Monod son fundamentales para la regulación celular en todos los organismos. La idea clave es que las proteínas no se sintetizan cuando no se las necesita. E. coli ahorra recursos celulares y energía evitando sintetizar las tres proteínas Lac cuando no hay necesidad de metabolizar lactosa, como por ejemplo, cuando otros azúcares como la glucosa, están presentes. La pregunta clave era cómo controla E. coli ciertos genes en respuesta a sus necesidades metabólicas?
Durante la Segunda Guerra Mundial, Monod ensayaba los efectos de las combinaciones de azúcares como fuente de nutrientes para E. coli. Descubrió que las bacterias a las cuales se las cultivaba en un medio que contenía dos azúcares diferentes generalmente mostraban dos fases de crecimiento. Por ejemplo, si se les proveía glucosa y lactosa, la primera sería metabolizada en primer lugar (fase de crecimiento I, véase la figura 2), y luego la lactosa (fase de crecimiento II). A este fenómeno se lo denomina diauxía.

El metabolismo de la lactosa no ocurre durante la primera parte de la curva de crecimiento diáuxico porque la β-galactosidasa no se sintetiza cuando hay presentes tanto glucosa como lactosa en el medio.
La explicación a este hecho dependía de la caracterización de mutaciones adicionales que afectaran a otros genes lac que no fueran explicados por el modelo clásico. Se supo de otros dos genes, cya y crp, que están mapeados lejos de lac, y cuando se encuentran mutados, resultan expresados a niveles más bajos en presencia de IPTF, e incluso en una cepa mutante para el represor o el operador. El descubrimiento del AMP cíclico en 1957 (en células eucariotas), y una década más tarde en E. coli, llevó a la demostración de que los mutantes con uno de estos genes defectuosos podían recobrar su actividad por adición de AMP cíclico al medio.
El gen cya codifica para la adenilato ciclasa, la cual produce AMP cíclico. En un mutante para cya, la ausencia de AMP cíclico hace unas diez veces más lenta la expresión de los genes lacZYA. La adición de AMP cíclico corrige los bajos niveles de expresión de Lac característicos de los mutantes para cya. El segundo gen, crp, codifica para una proteína llamada catabolite activator protein (proteína activadora de catabolitos, o CAP, por sus siglas en inglés), o catabolite repressor protein (proteína represora de catabolitos, o CRP, por sus siglas en inglés). [Es destacable que casi cuarenta años después, diferentes genetistas utilicen términos diferentes para el mismo gen, según cómo sea su sentimiento hacia los dos grupos que competían por el descubrimiento original.]
Esta regulación dual causa que la enzimas involucradas en el metabolismo de la lactosa sean fabricadas en pequeñas cantidades en presencia tanto de glucosa como de lactosa (a veces llamada leaky expression, del inglés expresión con pérdidas) debido a la inhibición de la unión de LacI al operador por parte de la lactosa, pero a concentraciones de AMPc altas y en presencia de lactosa, siempre hay niveles altos de expresión (Fase II en la Figura 2). [Leaky expression] es necesaria, de modo tal de permitir el metabolismo de algo de glucosa luego de que la fuente de glucosa haya sido agotada, pero antes de que la expresión de lac haya sido completamente activada.
En resumen:
- En ausencia de lactosa, no hay producción de enzima Lac (LacI está unido al operador).
- Cuando hay lactosa presente, pero también hay una fuente de carbono preferida en el medio (como la glucosa), entonces solo una pequeña cantidad de enzima se produce (LacI no está unido al operador).
- Cuando la lactosa es la fuente preferida de carbono (por ejemplo, en ausencia de glucosa) AMPc-CAP se unen al promotor y la producción de la enzima Lac se maximiza.
Entonces, por qué hay un retraso entre las dos fases de crecimiento? En primer lugar, la proteína regulatoria CAP debe ensamblarse sobre el operón lac, lo cual se traduce en un incremento en la producción de ARNm de lac. A mayor cantidad de copias de ARNm de lac, mayor será la cantidad de copias (véase traducción) de LacZ (β-galactosidasa para metabolizar lactosa) y LacY (lactosa permeasa para el transporte de la lactosa hacia el interior de la célula). Luego de una pausa necesaria para incrementar el nivel de las enzimas metabólicas de lactosa, las bacterias entran en una nueva y rápida fase de crecimiento celular.
Dos enigmas de la represión de catabolitos relacionada con como el nivel de AMPc está en realidad acoplado a la presencia de glucosa y, en segundo lugar, por qué a las células debiera siquiera preocuparles. Luego de que la lactosa es clivada forma glucosa y galactosa (fácilmente conversible a glucosa). En términos metabólicos, la lactosa es tan buena fuente de carbono y energía como la glucosa. El nivel de AMPc está relacionado, no a la concentración de glucosa intercelular, sino a la velocidad de transporte de glucosa, la cual influencia la actividad de la adenilato ciclasa. (Además, el transporte de glucosa también lleva a la inhibición directa de la lactosa permeasa). Solamente es posible especular acerca de por qué E. coli trabaja de esta manera. Todas las bacterias entéricas fermentan glucosa, lo que sugiere que la encuentran frecuentemente. Es posible que una pequeña diferencia en la eficiencia del transporte o el metabolismo de la glucosa vs. lactosa haga ventajoso para las células el regular al operón lac de esta manera.[cita requerida]
Referencias
[editar]- ↑ Cooper, Geoffrey M; Hausman, Robert E. La célula. Marban. p. 256. ISBN 978-84-7101-811-3.
- ↑ Levine, Louis (1979). «9». Biología del gen. Barcelona: Ediciones Omega S.A. pp. 210-213, 238-240. ISBN 84-282-0551-5.
- ↑ Ayala, Francisco J.; Kiger, John A. (1984). «13». Genética moderna. Barcelona: Omega S.A. pp. 432-439. ISBN 84-282-0720-8.
- ↑ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Morgan, David; Raff, Martin; Roberts, K.; Walter, P. (2016). «7». Biología molecular de la célula (6.ª edición). Barcelona: Omega S.A. pp. 384-385. ISBN 978-84-282-1638-8.
Enlaces externos
[editar]- www.ucm.es - Catabolitos
- www.ugr.es - Regulación
- La secuencia de ADN del operón lac de E. coli, el ARNm de los genes lacZYA, y el gen lacI están disponibles en GenBank (ver).