Archivo:Coronavirus. SARS-CoV-2.png
Apariencia
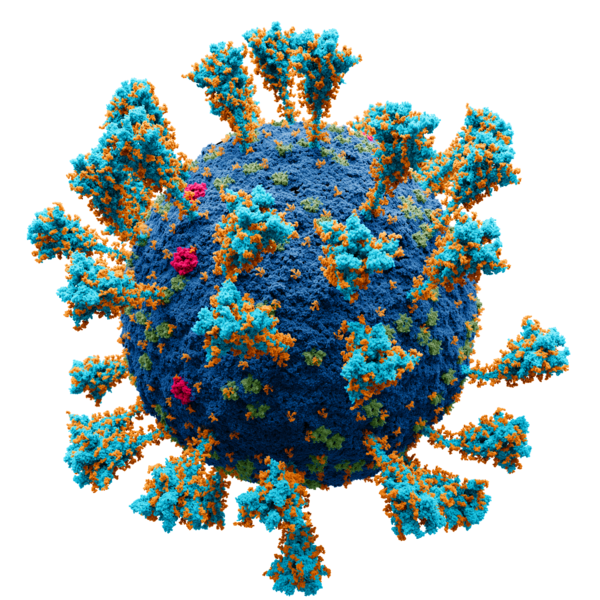
Tamaño de esta previsualización: 600 × 600 píxeles. Otras resoluciones: 240 × 240 píxeles · 480 × 480 píxeles · 768 × 768 píxeles · 1024 × 1024 píxeles · 2048 × 2048 píxeles.
Ver la imagen en su resolución original (2048 × 2048 píxeles; tamaño de archivo: 4,54 MB; tipo MIME: image/png)
Historial del archivo
Haz clic sobre una fecha y hora para ver el archivo tal como apareció en ese momento.
| Fecha y hora | Miniatura | Dimensiones | Usuario | Comentario | |
|---|---|---|---|---|---|
| actual | 22:17 9 ene 2022 |  | 2048 × 2048 (4,54 MB) | Jul059 | Lossless file size reduction |
| 03:58 24 sep 2021 |  | 2048 × 2048 (4,6 MB) | Iketsi | lossless compression | |
| 16:06 15 jun 2021 |  | 2048 × 2048 (5,34 MB) | AlexeySolodovnikov | fix color bug | |
| 14:28 13 jun 2021 |  | 2048 × 2048 (5,34 MB) | AlexeySolodovnikov | Мы обновили модель. В роли нашего научного консультанта выступил доктор биологических наук, специалист в области вирусологии, Никитин Н. А. и к.х.н специалист по молекулярному моделированию поверхностных вирусных белков Борисевич С.С. Под их руководством в модель были внесены следующие правки: Изменено количество S-белков с 90 до 38, количество M-белков было увеличено до 1000, а E-белков, как минорных компонентов мембраны, снижено до 15, HE-белок удалён. Также была принята во внимание шарни... | |
| 11:06 17 may 2021 |  | 2048 × 2048 (16,04 MB) | AlexeySolodovnikov | add alpha | |
| 18:41 4 may 2021 |  | 2048 × 2048 (16,04 MB) | AlexeySolodovnikov | Uploaded own work with UploadWizard |
Usos del archivo
Las siguientes páginas usan este archivo:
- B.1.1.523
- B.1.620
- Cronología de la pandemia de COVID-19 en enero de 2023
- SARS-CoV-2
- Subvariante Delta AY.4.2 del SARS-CoV-2
- Subvariantes delta del SARS-CoV-2
- Variante C.1.2 del SARS-CoV-2
- Variante XE del SARS-CoV-2
- Variante mu del SARS-CoV-2
- Variantes de SARS-CoV-2
- Usuario:Alejocat19/Taller/Anexo:Variantes del SARS-CoV-2
- Usuario:Alejocat19/Taller/Subvariante BA.2 del SARS-CoV-2
- Usuario:Alejocat19/Taller/Subvariante delta AY.20 del SARS-CoV-2
- Usuario:Alejocat19/Taller/Subvariantes ómicron del SARS-CoV-2
- Usuario:Alejocat19/Taller/Variante B.1.630 del SARS-CoV-2
- Usuario:Alejocat19/Taller/Variante eta del SARS-CoV-2
- Usuario:Gael Ángel
- Usuario:IntentandoArreglar
- Usuario:IntentandoArreglar/Coronavirus
- Usuario:IntentandoArreglar/FamiliarCoronavirus
- Usuario:IntentandoArreglar/FamiliarMuertoCorona
- Usuario:IntentandoArreglar/NoCoronavirus
- Usuario:IntentandoArreglar/UserboxesCreadas
- Usuario:Joseaperez/fuentes
- Usuario:Juan Pop Gamer
- Usuario:SixGael
- Usuario:Userbox/Salud
- Usuaria:Userbox mujer/Salud
- Usuario:Wawdanwaw
- Plantilla:Ficha de taxón/casos de prueba
- Anexo:Linajes y subvariantes del SARS-CoV-2
Uso global del archivo
Las wikis siguientes utilizan este archivo:
- Uso en alt.wikipedia.org
- Uso en ar.wikipedia.org
- مراكز السيطرة على الأمراض والوقاية منها
- فيروس كورونا
- مستخدم:Amira Hashem1996/ملعب
- مناطق انتشار جائحة فيروس كورونا حسب الدولة والمنطقة
- عزل ووهان 2020
- قائمة حوادث كراهية الأجانب والعنصرية المرتبطة بجائحة فيروس كورونا
- مستشفى هوو شين شان
- مستشفى لي شين شان
- جائحة فيروس كورونا في العراق
- معهد ووهان لأبحاث الفيروسات
- جائحة فيروس كورونا في إيطاليا
- جائحة فيروس كورونا في الجزائر
- جائحة فيروس كورونا في اليونان
- اللجنة الوطنية للصحة (الصين)
- جائحة فيروس كورونا في الكويت
- جائحة فيروس كورونا في الكاميرون
- المركز الصيني لمكافحة الأمراض والوقاية منها
- جائحة فيروس كورونا في البوسنة والهرسك
- أثر جائحة فيروس كورونا على الحياة الاجتماعية
- مستشفى ووهان المركزي
- جائحة فيروس كورونا في الأردن
- أثر جائحة فيروس كورونا على الرياضة
- جائحة فيروس كورونا في السودان
- جائحة فيروس كورونا في فرنسا
- جائحة فيروس كورونا في إفريقيا
- جائحة فيروس كورونا في جمهورية الكونغو الديمقراطية
- جائحة فيروس كورونا في الغابون
- انهيار فندق شينجيا إكسبريس
- جائحة فيروس كورونا في توغو
- جائحة فيروس كورونا في غينيا
- جائحة فيروس كورونا في رواندا
- جائحة فيروس كورونا في ساحل العاج
- جائحة فيروس كورونا في ناميبيا
- جائحة فيروس كورونا في كينيا
- جائحة فيروس كورونا في مايوت
- جائحة فيروس كورونا في لا ريونيون
- قيود السفر بسبب جائحة فيروس كورونا
- جائحة فيروس كورونا في غينيا الاستوائية
- جائحة فيروس كورونا في جمهورية إفريقيا الوسطى
- جائحة فيروس كورونا في جمهورية الكونغو
- جائحة فيروس كورونا في سيشل
- جائحة فيروس كورونا في ليبيريا
- جائحة فيروس كورونا في الصومال
- جائحة فيروس كورونا في تنزانيا
- جائحة فيروس كورونا في كازاخستان
- جائحة فيروس كورونا في أوروبا
- لقاح كوفيد-19
- جائحة فيروس كورونا في أوقيانوسيا
- جائحة فيروس كورونا في كولومبيا
Ver más uso global de este archivo.




