Complementariedad (biología molecular)
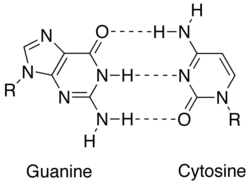

En biología molecular, la complementariedad describe una relación entre dos estructuras cada una siguiendo el principio de bloqueo y clave. En la naturaleza, la complementariedad es el principio básico de la replicación y transcripción del ADN, ya que es una propiedad compartida entre dos secuencias de ADN o ARN, de manera que cuando están alineadas antiparalelas entre sí, las bases de nucleótidos en cada posición de las secuencias serán complementarias, Como mirar en el espejo y ver el revés de las cosas. Este emparejamiento de base complementario permite a las células copiar información de una generación a otra e incluso encontrar y reparar daños a la información almacenada en las secuencias. El grado de complementariedad entre dos cadenas de ácido nucleico puede variar, desde la complementariedad completa (cada nucleótido es a través de su opuesto) hasta no complementariedad (cada nucleótido no es a través de su opuesto) y determina la estabilidad de las secuencias para estar juntos. Además, varias funciones de reparación del ADN, así como funciones reguladoras, se basan en la complementariedad de pares de bases. En biotecnología, el principio de complementariedad de pares de bases permite la generación de híbridos de ADN entre ARN y ADN, y abre la puerta a herramientas modernas como las bibliotecas de ADNc. Aunque la mayor parte de la complementariedad se ve entre dos cadenas separadas de ADN o ARN, también es posible que una secuencia tenga complementariedad interna resultando en la secuencia uniéndose a sí misma en una configuración plegada.
Complementariedad de pares de bases de ADN y ARN
[editar]

| Ácido nucleico | Nucleobases | La base complementa |
| ADN | Adenina(A), timina(T), guanina(G), citosina(C) | A=T, G≡C |
| ARN | Adenina(A), uracilo(U), guanina(G), citosina(C) | A=U, G≡C |
Autocomplementariedad y bucles de horquilla
[editar]

La autocomplementariedad se refiere al hecho de que una secuencia de ADN o ARN puede plegarse sobre sí misma, creando una estructura de doble hebra. Dependiendo de lo cerca que estén las partes de la secuencia que son auto-complementarias, la hebra puede formar bucles horquilla, uniones, protuberancias o bucles internos.[1] Es más probable que el ARN forme este tipo de estructuras debido a la unión de pares de bases no observada en el ADN, como la unión de guanina con uracilo.[1]
Bioinformática
[editar]La complementariedad permite que la información contenida en el ADN o el ARN se almacene en una sola hebra. La cadena complementaria se puede determinar a partir de la plantilla y viceversa como en las bibliotecas de cDNA. Esto también permite el análisis, como la comparación de las secuencias de dos especies diferentes. Las abreviaturas se han desarrollado para escribir secuencias cuando hay desajustes (códigos de ambigüedad) o para acelerar la lectura de la secuencia opuesta en el complemento (ambigramas).
Ambigramas
[editar]Se pueden usar caracteres específicos para crear una notación de ácido nucleico (ambigráfico) adecuada para bases complementarias (es decir, guanina = b, citosina = q, adenina = n, y timina = u), lo que hace posible complementar secuencias completas de ADN simplemente Girando el texto "al revés".[2] Por ejemplo, con el alfabeto anterior, buqn (GTCA) se leería como ubnq (TGAC, complemento inverso) si se pone al revés.
- qqubqnnquunbbqnbb
- bbnqbuubnnuqqbuqq
Las notaciones ambigráficas visualizan fácilmente tramos de ácidos nucleicos complementarios tales como secuencias palindrómicas.[3] Esta característica se mejora cuando se utilizan fuentes o símbolos personalizados en lugar de caracteres ASCII o incluso Unicode comunes.[3]

Véase también
[editar]Referencias
[editar]- ↑ a b Watson, James, Cold Spring Harbor Laboratory, Tania A. Baker, Massachusetts Institute of Technology, Stephen P. Bell, Massachusetts Institute of Technology, Alexander Gann, Cold Spring Harbor Laboratory, Michael Levine, University of California, Berkeley, Richard Losik, Harvard University ; with Stephen C. Harrison, Harvard Medical. Molecular biology of the gene (Seventh edition. edición). Boston: Benjamin-Cummings Publishing Company. ISBN 978-0-32176243-6.
- ↑ Rozak DA (2006). «The practical and pedagogical advantages of an ambigraphic nucleic acid notation». Nucleosides Nucleotides Nucleic Acids 25 (7): 807-13. PMID 16898419. doi:10.1080/15257770600726109.
- ↑ a b Rozak, DA; Rozak, AJ (mayo de 2008). «Simplicity, function, and legibility in an enhanced ambigraphic nucleic acid notation.». BioTechniques 44 (6): 811-3. PMID 18476835. doi:10.2144/000112727.
