Ácido nucleico bloqueado
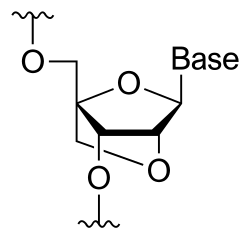
Un ácido nucleico bloqueado (LNA), también conocido como ADN inaccesible, es un nucleótido de ADN modificado. Los restos de ribosa en un nucleótido de LNA se encuentra modificado con un puente extra que conecta el oxígeno 2' con el carbono 4'. El puente bloquea la conformación de la ribosa en una configuración 3'-endo (norte), la cual se encuentra a menudo en la forma de duplos en forma de A. Los nucleótidos LNA pueden ser mezclados con residuos de ADN o ARN en la secuencia del oligonucleótido allí donde se desee. Algunos oligómeros son químicamente sintetizados y se encuentran disponibles comercialmente. La conformación de la ribosa bloqueada mejora las propiedades de apilamiento de bases y la preorganización del esqueleto de azúcar fosfato. Esto aumenta significativamente las propiedades de hibridación del oligonucleótido tales como la temperatura de desnaturalización.[1]
Los ANB fueron sintetizados en forma independiente por el grupo de Jesper Wengel[2] en 1998, siendo sintetizados por primera vez por el grupo de Takeshi Imanishi[3] en 1997. Los derechos intelectuales concernientes a los ANB fueron asegurados en 1997 por la compañía de biotecnología danesa Exigon A/S.[4]

Los nucleótidos ANB son utilizados para incrementar la sensibilidad y la especificidad de expresión en los microarreglos de ADN, sondas FISH fluorescentes para técnicas de hibridación in situ, sondas para PCR en tiempo real y otras técnicas de biología molecular basadas en oligonucleótidos. Por el momento la única técnica eficiente para la detección in situ de micro ARN es el uso de ANBs. Un triplete de nucleótidos ANB rodeando un sitio único de emparejamiento defectuoso maximiza la especificidad de una sonda ANB, a menos que la sonda contenga la base guanina de la pareja G-T mal acoplada.[5]
La utilización de oligonucleótidos basados en LNA como herramientas terapéuticas es un campo emergente en biotecnología. La compañía farmacéutica danesa Santaris Pharma a/s es propietaria de todos los derechos por la utilización terapéutica de la tecnología de los ANBs,[6] y actualmente se encuentra desarrollando una nueva droga para el tratamiento de la Hepatitis C basada en los ANB llamada miravasen, esta droga hace diana sobre el gen miR-122, a fines de 2010 esta droga se encontraba en la fase II de pruebas clínicas.[7]
Beneficios de la tecnología de los LNA[editar]
Algunos de los beneficios que incluye la utilización de LNAs son:
- Resultan ideales para la detección de secuencias cortas de ADN y ARN
- Incrementa la estabilidad térmica de los ácidos nucleicos de doble cadena
- Son capaces de discriminar entre ácidos nucleicos con un nucleótido de diferencia
- Son resistentes a las exo y endonucleasas lo que les otorga una alta estabilidad en aplicaciones in vivo e in vitro
- Alta especificidad en el reconocimiento de secuencias diana
- Facilitan la normalización de la temperatura de desnaturalización
- La capacidad de invadir emparejamientos de alta estabilidad les permite detectar incluso muestras con secuencias de difícil acceso.
- Resultan compatibles con los procesos enzimáticos estandarizados[8]
Aplicaciones de la tecnología de LNAs[editar]
Algunas de las aplicaciones de los LNAs incluyen:
- PCR específica de alelos: permiten el diseño de secuencias iniciadores más cortas, sin comprometer la capacidad de unión al sito específico[9]
- Permite generar perfiles de expresión genética en microarreglos, aumentando la sensibilidad y la selectividad con cantidades más pequeñas de sustratos[10]
- Investigación de ARN de pequeño tamaño
- Genotipificación de SNPs
- Producción de oligonucleótidos de ARNm antisentido
- ARNi
- ADNzimas
- Sondas de polarización fluorescente
- Balizas moleculares
- Reparación de genes y salto de exones
- Detección de variantes de splicing
- Hibridación comparativa de genomas (GCH)[11]
Existen otras aplicaciones terapéuticas y diagnósticas que se encuentran actualmente en desarrollo.[11]
Referencias[editar]
- ↑ Kaur, H., H; Arora, A; Wengel, J; Maiti, S (2006). «Thermodynamic, Counterion, and Hydration Effects for the Incorporation of Locked Nucleic Acid Nucleotides into DNA Duplexes». Biochemistry 45 (23): 7347-55. PMID 16752924. doi:10.1021/bi060307w.
|apellido1=y|autor=redundantes (ayuda);|autor2=y|apellido2=redundantes (ayuda);|autor3=y|apellido3=redundantes (ayuda);|autor4=y|apellido4=redundantes (ayuda) - ↑ Alexei A. Koshkin; Sanjay K. Singh, Poul Nielsen, Vivek K. Rajwanshi, Ravindra Kumar, Michael Meldgaard, Carl Erik Olsen, Jesper Wengel (1998). «LNA (Locked Nucleic Acids): Synthesis of the adenine, cytosine, guanine, 5-methylcytosine, thymine and uracil bicyclonucleoside monomers, oligomerisation, and unprecedented nucleic acid recognition». Tetrahedron 54 (14): 3607-30. doi:10.1016/S0040-4020(98)00094-5.
- ↑ Satoshi Obika; Daishu Nanbu, Yoshiyuki Hari, Ken-ichiro Morio, Yasuko In, Toshimasa Ishida, Takeshi Imanishi (1997). «Synthesis of 2′-O,4′-C-methyleneuridine and -cytidine. Novel bicyclic nucleosides having a fixed C3'-endo sugar puckering». Tetrahedron Lett. 38 (50): 8735-8. doi:10.1016/S0040-4039(97)10322-7.
- ↑ «Copia archivada». Archivado desde el original el 3 de enero de 2011. Consultado el 22 de junio de 2012.
- ↑ You Y.; Moreira B.G.; Behlke M.A. and Owczarzy R. (2006). «Design of LNA probes that improve mismatch discrimination». Nucleic Acids Res. 34 (8): e60. PMC 1456327. PMID 16670427. doi:10.1093/nar/gkl175.
- ↑ «Developing LNA technology for new-generation cancer drugs» (PDF). SP2 Magazine. March.
- ↑ Franciscus, Alan (2010). «Hepatitis C Treatments in Current Development». HCV Advocate. Archivado desde el original el 29 de septiembre de 2007.
- ↑ «Locked Nucleic Acid Technology». Archivado desde el original el 20 de agosto de 2009. Consultado el 22 de junio de 2012.
- ↑ Bonetta, Laura (2005). «Prime time for real-time PCR». Nat. Methods 2 (4): 305-312. doi:10.1038/nmeth0405-305.
- ↑ Roberts, Peter (2006). «MicroRNA expression profiling on arrays enhanced with locked nucleic acids». Nat. Methods 3 (4). doi:10.1038/nmeth869.
- ↑ a b «Copia archivada». Archivado desde el original el 1 de septiembre de 2010. Consultado el 22 de junio de 2012.
<ref> con nombre «Franciscus» definida en el grupo «» para <references> no tiene contenido.Enlaces externos[editar]
- LNA Oligo Tools and Design Guidelines Archivado el 20 de agosto de 2009 en Wayback Machine.
- LNA Oligo melting temperature including mismatches
- LNA Summary
- Vester B, Wengel J (octubre de 2004). «LNA (locked nucleic acid): high-affinity targeting of complementary RNA and DNA». Biochemistry 43 (42): 13233-41. PMID 15491130. doi:10.1021/bi0485732.
- Petersen M, Wengel J (febrero de 2003). «LNA: a versatile tool for therapeutics and genomics». Trends Biotechnol. 21 (2): 74-81. PMID 12573856. doi:10.1016/S0167-7799(02)00038-0.
- Ng PS, Bergstrom DE (enero de 2005). «Alternative nucleic acid analogues for programmable assembly: hybridization of LNA to PNA». Nano Lett. 5 (1): 107-11. PMID 15792422. doi:10.1021/nl048246f.
