BioPerl
| BioPerl | ||
|---|---|---|
| Archivo:BioPerlLogo.png | ||
| Información general | ||
| Tipo de programa | Bioinformática | |
| Lanzamiento inicial | 11 de junio de 2002 | |
| Licencia | Licencia artística y GPL | |
| Información técnica | ||
| Programado en | Perl | |
| Versiones | ||
| Última versión estable | 1.6.9 ( 14 de abril de 2011 (13 años y 26 días)) | |
| Última versión en pruebas | Nightly builds () | |
| Enlaces | ||
BioPerl es un software de código abierto diseñado a partir de la colaboración entre bioinformáticos, biólogos y científicos del área de la computación, ante la necesidad de analizar y resolver problemas de la bioinformática.[1] BioPerl es un proyecto activo financiado por la Open Bioinformatics Foundation, basado en módulos de Perl con el fin de facilitar la administración y manipulación de información relacionada con ciencias de la vida .[1] Tales módulos son interfaces de tipos de datos de secuencias, alineamientos (BLAST, Clustal), características y localizaciones génicas y bases de datos (GenBank), entre otras. Con el fin de aprovechar BioPerl, el usuario necesita una comprensión básica del lenguaje de programación Perl que incluya una comprensión del uso de referencias, módulos, objetos y métodos.
La primera versión estable fue lanzada el 11 de junio de 2002; la última estable (en términos de la API) es la 1.6.9, lanzada en abril de 2011. También hay lanzamientos periódicos producidos por desarrolladores. La versión 1.6.0 es considerada la más estable (en términos de errores) y la versión de BioPerl se recomienda para el uso diario, se basa en Nightly Builds, también estable.
BioPerl ha desempeñado un papel integral en el Proyecto Genoma Humano.[2]
Características[editar]
BioPerl provee de módulos de software para muchas tareas típicas de programación en bioinformática. Estas incluyen:
- Acceso a datos a secuencias de ADN y de aminoácidos de bases de datos locales y remotas.
- Transformación de formatos de bases de datos y archivos de registro.
- Manipulación de secuencias individuales.
- Búsqueda de secuencias similares.
- Creación y manipulación de alineamientos de secuencias.
- Búsqueda de genes y otras estructuras del ADN genómico.
- Desarrollo de anotaciones de secuencia legibles por máquina.
Aplicaciones[editar]
Además de ser usado directamente por usuarios finales,[3] BioPerl también ha provisto de una base para una variedad de herramientas bioinformáticas, incluyendo entre otras:
- Trinity[4]
- SynBrowse[5]
- GeneComber[6]
- TFBS[7]
- MIMOX[8]
- BioParser[9]
- Diseño de cebadores degenerados[10]
- Búsqueda en bases de datos públicas[11]
- Current Comparative Table[12]
Nuevas herramientas y algoritmos de desarrolladores externos son con frecuencia integrados directamente en BioPerl:
- Manejo de árboles filogenéticos y taxones anidados[13]
- Herramientas web FPC[14]
Instalación en UNIX[editar]
Requerimientos del Sistema[editar]
- Perl version 5.6.1 o superior. Se recomienda Perl version 5.8 o superior.
- External modules: BioPerl usa funciones provistas en otros módulos de Perl. Algunas de estas funciones están incluidas en el paquete estándar de Perl y otras se pueden descargar desde CPAN y son instaladas automáticamente si se instala BioPerl de manera fácil. Esta lista de módulos externos se puede encontrar en la página oficial de BioPerl.
Módulos más importantes[editar]
| Módulos | Descripción |
|---|---|
| Bio::Seq | Secuencias y sus propiedades |
| Bio::SeqIO | Entrada/Salida de datos de secuencias |
| Bio::Index | Indexación de archivos de secuencias |
| Bio::DB | Acceso remoto a bases de datos de secuencias y referencias por medio de conexión HTTP |
| Bio::Seq::Feature | Anotaciones y características que tiene la ubicación de una secuencia |
| Bio::Annotation | Anotaciones genéricas: comentarios y referencias |
| Bio::AlignIO, Bio::SimpleAlign | Alineamientos múltiples de secuencias |
| Bio::Search, Bio::SearchIO | Búsqueda en bases de datos de secuencias |
| Bio::Map, Bio::MapIO | Mapas Biológicos |
| Bio::LiveSeq, Bio::Variation | Mutaciones y variaciones de secuencia |
| Bio::Tree, Bio::Tree::IO | Árboles Filogenéticos |
| Bio::Structure | Datos estructurales de proteínas |
| Bio::Graphics | Visualización de secuencias |
Ejemplo de lectura de datos de secuencias[editar]
Los módulos IO (entrada/salida) leen datos formateados y crean representaciones en memoria de los datos llamados objetos.
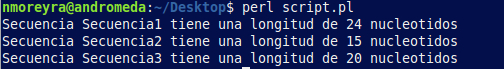
#!/usr/bin/perl -w
use strict;
use Bio::SeqIO;
my $seqio = Bio::SeqIO->new(-format => "fasta", -file => "secuencias.fasta");
while(my $secuencia = $seqio->next_seq()){
print "Secuencia ", $secuencia->display_id, " tiene una longitud de ",
$secuencia->length, " nucleotidos\n";
}
Referencias[editar]
- ↑ a b Stajich, Jason E. (2002). «The Bioperl Toolkit: Perl Modules for the Life Sciences». Genome Research. PMID 12368254. doi:10.1101/gr.361602. Consultado el 4 de diciembre de 2015.
- ↑ Lincoln D. Stein (1996). «How Perl saved the Human Genome Project». The Perl Journal 1 (2). Archivado desde el original el 2 de febrero de 2007.
- ↑ Khaja R, MacDonald J, Zhang J, Scherer S. «Methods for identifying and mapping recent segmental and gene duplications in eukaryotic genomes». Methods Mol Biol 338: 9-20. PMID 16888347.
- ↑ Grabherr, Manfred (15 de mayo de 2015). «Full-length transcriptome assembly from RNA-Seq data without a reference genome». Nature Biotechnology. PMID 21572440. doi:10.1038/nbt.1883. Consultado el 4 de diciembre de 2015.
- ↑ Pan X, Stein L, Brendel V (2005). «SynBrowse: a synteny browser for comparative sequence analysis». Bioinformatics 21 (17): 3461-8. PMID 15994196. doi:10.1093/bioinformatics/bti555.
- ↑ Shah S, McVicker G, Mackworth A, Rogic S, Ouellette B (2003). «GeneComber: combining outputs of gene prediction programs for improved results». Bioinformatics 19 (10): 1296-7. PMID 12835277. doi:10.1093/bioinformatics/btg139.
- ↑ Lenhard B, Wasserman W (2002). «TFBS: Computational framework for transcription factor binding site analysis». Bioinformatics 18 (8): 1135-6. PMID 12176838. doi:10.1093/bioinformatics/18.8.1135.
- ↑ Huang J, Gutteridge A, Honda W, Kanehisa M (2006). «MIMOX: a web tool for phage display based epitope mapping». BMC Bioinformatics 7: 451. PMID 17038191. doi:10.1186/1471-2105-7-451.
- ↑ Catanho M, Mascarenhas D, Degrave W, de Miranda A (2006). «BioParser: a tool for processing of sequence similarity analysis reports». Appl Bioinformatics 5 (1): 49-53. PMID 16539538. doi:10.2165/00822942-200605010-00007.
- ↑ Wei X, Kuhn D, Narasimhan G. «Degenerate primer design via clustering». Proc IEEE Comput Soc Bioinform Conf 2: 75-83. PMID 16452781.
- ↑ Croce O, Lamarre M, Christen R (2006). «Querying the public databases for sequences using complex keywords contained in the feature lines». BMC Bioinformatics 7: 45. PMID 16441875. doi:10.1186/1471-2105-7-45.
- ↑ Landsteiner B, Olson M, Rutherford R (2005). «Current Comparative Table (CCT) automates customized searches of dynamic biological databases». Nucleic Acids Res 33 (Web Server issue): W770-3. PMID 15980582. doi:10.1093/nar/gki432.
- ↑ Llabrés M, Rocha J, Rosselló F, Valiente G (2006). «On the ancestral compatibility of two phylogenetic trees with nested taxa». J Math Biol 53 (3): 340-64. PMID 16823581. doi:10.1007/s00285-006-0011-4.
- ↑ Pampanwar V, Engler F, Hatfield J, Blundy S, Gupta G, Soderlund C (2005). «FPC Web tools for rice, maize, and distribution». Plant Physiol 138 (1): 116-26. PMID 15888684. doi:10.1104/pp.104.056291.
