LRRK1
| serina/treonina-proteína quinasa 1 rica en leucina LRRK1 | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB |
Buscar ortólogos: | |||
| Identificadores | ||||
| Nomenclatura |
Otros nombres leucine-rich repeat kinase 1
FLJ23119 KIAA1790 ROCO1 RIPK6 | |||
| Símbolo | LRRK1 (HGNC: 18608) | |||
| Identificadores externos |
Bases de datos de enzimas
| |||
| Número EC | 2.7.11.1 | |||
| Locus | Cr. 15 q26.3 | |||
| Estructura/Función proteica | ||||
| Tamaño | 2015 (aminoácidos) | |||
| Peso molecular | 225.393 (Da) | |||
| Funciones | kinase | |||
| Dominio proteico | Roc, COR, quinasa y WD40 | |||
| Motivos | AlphaFold | |||
| UniProt |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
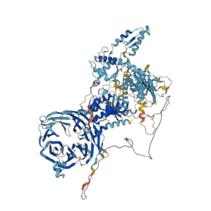
La serina/treonina-proteína quinasa 1 rica en leucina es una enzima que está codificada por el gen LRRK1[1][2][3]y que pertenece a la familia de proteínas ROCO en humanos. Es un gen que codifica una proteína de múltiples dominios, que es una quinasa repetida rica en leucina y una proteína de unión a GDP o GTP.[4] A pesar de ser un homólogo de LRRK2, también perteneciente a la familia ROCO, es menos estudiada y tiene un nivel adicional de autoinhibición que previene la activación de la quinasa.
Su mutación provoca la pérdida de función autonómica recesiva[5]y tiene un papel fundamental en la regulación de la organización del citoesqueleto, actividad de osteoclastos y resorción ósea. Además, también tiene un papel menor en los parámetros de formación de huesos. Asimismo, puede servir como tratamiento farmacológico para tratar la osteoporosis y otras enfermedades óseas.[4]
Función[editar]
La proteína tiene acción serina/treonina-proteína quinasa y posee el número (EC:2.7.11.1).
El gen LRRK1 sintetiza una proteína que pertenece a la familia de proteínas Roco, de la cual se conocen 4 proteínas en humanos y vertebrados: MASL1, DAPK1, LRRK1 y LRRK2.[6] Estas cuatro proteínas tienen funciones distintas, aunque algunas no están muy claras debido a que no han sido estudiadas en profundidad.
- Aun así, se sabe que DAPK1 está involucrada en el control de la apoptosis, ya que su inhibición lleva a la supresión de esta;[7] y que LRRK2 está involucrada en la enfermedad de Párkinson. De las otras dos proteínas, MASL1 y LRRK1, no se conoce su función exacta, aunque hay estudios que sugieren que MASL1 está involucrada en la transducción de señales y en el control del crecimiento celular en humanos.[8] [9]
Desafortunadamente, el rol fisiológico y patológico exacto de LRRK1 permanece desconocido, aunque en los últimos años, varios estudios han descubierto que tiene un rol importante en la regulación de la masa ósea en los humanos.[10] En concreto, en 2016 un estudio descubrió el papel del gen LRRK1 en la regulación del crecimiento óseo mediante un experimento con ratones con una deficiencia de este gen. Este estudio concluyó que la falta de LRRK1 puede causar osteopetrosis severa, y la sobre expresión de este gen en células HEK 293 se ha descubierto que induce la proliferación de células.[4]
Recientemente, un estudio japonés descubrió que la proteína LRRK1 está involucrada en la mitofagia mediada por la proteína Parkin, un proceso mediante el cual se degradan mitocondrias por autofagia. El complejo ULK interactúa con ATG13A3, una proteína relacionada con la autofagia, para incorporar LRRK1 e inicia la mitofagia mediante la fosforilación de Rab7 en su serina 72.[11]
Estructura[editar]
Se ha observado que la proteína LRRK1 se puede encontrar, en disolución acuosa, tanto en forma de monómero como en forma de dímero y que no hay una preferencia ante ninguna de estas especies.[12]La estructura general que presenta el monómero es compacta, tiene forma de "O" y son observables los dominios Roc, COR, quinasa y WD40 agrupados en forma de "J". Los dominios Roc-COR son unidos con los WD40 mediante puentes ricos en leucina aunque no forman un contacto directo. Se encuentra también un motivo Switch-I GTPasa el cual se encuentra en una conformación estable con el enlace de una molécula de GTP.[13]
La proteína LRRK1 contiene repeticiones ANK N-terminales. Se ha hecho predicciones tridimensionales de esta proteína y se he observado cierta similitud entre los dominios ANK de LRRK1 y de la estructura secundaria de la repetición ANK, ya que ambas contienen un dominio SH3 y una región rica en prolina que permite regular la apoptosis celular.[4]
Diferencias entre las proteínas LRRK1 y LRRK2[editar]
Es importante resaltar la diferencia en la posición de los LRR que se halla entre las proteínas LRRK1 y LRRK2.
En la proteína LRRK2, los grupos LRR junto con los dominios ANK cubren el dominio quinasa, de modo que permiten una regulación al acceso del sustrato. Esta acción implica el reordenamiento de los extremos N-terminales y de los grupos LRR. Por otro lado, en la proteína LRRK1, los grupos LRR no forman interfaces con el resto de la proteína, con lo que no hay una obstrucción del dominio quinasa.[12] Además, el grupo Roc, en la proteína LRRK2 está desplazado unos 10Å (respecto la proteína LRRK1) de modo que hay una adaptación más compacta de los grupos LRR en LRRK2.[12]
El dominio quinasa tiene una estructura parecida en ambas proteína. Sin embargo, en la proteína LRRK1 aparece una hélice αC.[12]

Estas diferencias estructurales conducen a pensar que ambas proteínas ejerzan distintas funciones.[4]
Andamios estructurales[editar]
Existen andamios estructurales[14]que permiten unir los dominios COR-B y quinasa, así como regular la actividad del dominio quinasa[15] mediante la activación e inactivación de este dominio. Uno de estos andamios estructurales es la alargada hélice αC que une el lóbulo N de la quinasa con el COR-B. Esta hélice αC cambiará de la posición "dentro" o posición "fuera" tras la activación de este dominio, permitiendo así una reestructuración del núcleo de la quinasa para permitir la fosforilación del sustrato.[16]
Otro de estos andamios estructurales es la hélice DK colocada en un ángulo de 30o con respecto la hélice αC (como se observa en la imagen) y forma contactos electroestáticos extensos con ella. Los residuos hidrofóbicos que se generan en la hélice DK se empaquetan con el dominio COR-B, de modo que queda bien estabilizado.[12]
Patologías asociadas[editar]
Aunque la proteína LRRK1 no ha sido tan estudiada como su homóloga LRRK2, diferentes estudios han demostrado su implicación en la regulación de la organización del citoesqueleto y en diversos trastornos óseos.[4]
Una de las principales enfermedades que se deben al mal funcionamiento de esta proteína es la osteopetrosis. La principal característica de esta es que los huesos son demasiado densos ya que el número de osteoclastos es anormalmente reducido. Debido a varias investigaciones, se pudo identificar que los organismos que mostraban alteraciones en la producción de la proteína LRRK1 presentaban osteopetrosis severa y una notable reducción en la resorción ósea.[17]Posteriormente, se ha descubierto que la proteína controla la actividad de estas células mediante el control del tráfico y la secreción lisosomal.[18]
Asimismo, se ha descubierto que una mutación en el gen LRRK1 y la actividad osteoclástica anormal es la causante de la displasia metafisiaria osteosclerótica. Esta enfermedad está caracterizada por la osteosclerosis localizada principalmente en la metáfisis de los huesos largos.[19]
Por otro lado, también se ha encontrado una relación entre la proteína LRRK1 y la correcta activación de los linfocitos B y por ende, en la respuesta humoral. Debido a unos experimentos, se pudo descubrir que los ratones que tenían un mal funcionamiento de la proteína LRRK1 presentaban alteraciones en el desarrollo de las células B y en la producción de anticuerpos. Estos animales eran incapaces de producir la inmunoglobulina E y por ello la supervivencia y la capacidad de proliferación disminuían considerablemente.[20]
Referencias[editar]
- ↑ Nagase, T. (2001). «Prediction of the Coding Sequences of Unidentified Human Genes. XX. The Complete Sequences of 100 New cDNA Clones from Brain Which Code for Large Proteins in vitro». DNA Research (en inglés) 8 (2): 85-95. ISSN 1340-2838. doi:10.1093/dnares/8.2.85. Consultado el 22 de julio de 2020.
- ↑ Bosgraaf, Leonard; Van Haastert, Peter J.M. (2003). «Roc, a Ras/GTPase domain in complex proteins». Biochimica et Biophysica Acta (BBA) - Molecular Cell Research (en inglés) 1643 (1-3): 5-10. doi:10.1016/j.bbamcr.2003.08.008. Consultado el 22 de julio de 2020.
- ↑ "Entrez Gene: LRRK1 leucine-rich repeat kinase 1"
- ↑ a b c d e f R. Xing, Weirong; Goodluck, Helen; Zeng, Canjun; Mohan, Subburaman (2017). «Role and mechanism of action of leucine-rich repeat kinase 1 in bone». PubMed Central. doi:10.1038/boneres.2017.3. Consultado el 14 de octubre de 2023.
- ↑ . M. Reimer, Janice; M. Dickey, Andrea; Lin, Yu Xuan; G. Abrisch, Robert; Mathea, Sebastian;Chaterjee, Deep; et al. (2022). «Structure of LRRK1 and mechanisms of autoinhibition and activation» [Estructura de LRRK1 y mecanismos de autoinhibición y activación]. doi:10.1101/2022.11.22.517582. Consultado el 12 de octubre de 2023.
- ↑ Marín, Ignacio; Egmond, Wouter N.; Haastert, Peter J. M. (2008). «The Roco protein family: a functional perspective». The FASEB Journal (en inglés) 22 (9): 3103-3110. ISSN 0892-6638. doi:10.1096/fj.08-111310. Consultado el 14 de octubre de 2023.
- ↑ Cohen, Ofer; Feinstein, Elena; Kimchi, Adi (1997). «DAP-kinase is a Ca2+/calmodulin-dependent, cytoskeletal-associated protein kinase, with cell death-inducing functions that depend on its catalytic activity». The EMBO Journal 16 (5): 998-1008. doi:10.1093/emboj/16.5.998. Consultado el 22 de octubre de 2023.
- ↑ Shinomiya, Takashi; Mori, Toshiki; Ariyama, Yoji; Sakabe, Tomoya; Fukuda, Yoji; Murakami, Yasushi; et al. (1999). <337::aid-gcc7>3.0.co;2-o «Comparative genomic hybridization of squamous cell carcinoma of the esophagus: The possible involvement of theDP1 gene in the 13q34 amplicon». Genes, Chromosomes and Cancer 24 (4): 337-344. ISSN 1045-2257. doi:10.1002/(sici)1098-2264(199904)24:4<337::aid-gcc7>3.0.co;2-o. Consultado el 22 de octubre de 2023.
- ↑ Lewis, Patrick A. (2009). «The function of ROCO proteins in health and disease». Biology of the Cell (en inglés) 101 (3): 183-191. ISSN 0248-4900. doi:10.1042/BC20080053. Consultado el 22 de octubre de 2023.
- ↑ Iida, Aritoshi; Xing, Weirong; Docx, Martine K F.; Nakashima, Tomoki; Wang, Zheng; Kimizuka, Mamori; et al. (2016). «Identification of biallelic LRRK1 mutations in osteosclerotic metaphyseal dysplasia and evidence for locus heterogeneity». Journal of Medical Genetics (en inglés) 53 (8): 568-574. ISSN 0022-2593. doi:10.1136/jmedgenet-2016-103756. Consultado el 14 de octubre de 2023.
- ↑ Fujita, Keitaro; Kedashiro, Shin; Yagi, Takuya; Hisamoto, Naoki; Matsumoto, Kunihiro; Hanafusa, Hiroshi (2022). «The ULK complex–LRRK1 axis regulates Parkin-mediated mitophagy via Rab7 Ser-72 phosphorylation». Journal of Cell Science (en inglés) 135 (23). ISSN 0021-9533. doi:10.1242/jcs.260395. Consultado el 14 de octubre de 2023.
- ↑ a b c d e Metcalfe, Riley D.; Martinez Fiesco, Juliana A.; Bonet-Ponce, Luis; Kluss, Jillian H.; Cookson, Mark R.; Zhang, Ping (2023). «Structure and regulation of full-length human leucine-rich repeat kinase 1». Nature Communications 14: 4797. ISSN 2041-1723. PMID 37558661. doi:10.1038/s41467-023-40532-2. Consultado el 14 de octubre de 2023.
- ↑ Vetter, I. R.; Wittinghofer, A. (2001). «The guanine nucleotide-binding switch in three dimensions». Science (New York, N.Y.) 294 (5545): 1299-1304. ISSN 0036-8075. PMID 11701921. doi:10.1126/science.1062023. Consultado el 14 de octubre de 2023.
- ↑ Garbett, Damien; Bretscher, Anthony (2014). «The surprising dynamics of scaffolding proteins». Molecular Biology of the Cell 25 (16): 2315-2319. ISSN 1059-1524. PMC 4142605. PMID 25122925. doi:10.1091/mbc.E14-04-0878. Consultado el 14 de octubre de 2023.
- ↑ Taylor, Susan S.; Wu, Jian; Bruystens, Jessica G.H.; Del Rio, Jason C.; Lu, Tsan-Wen; Kornev, Alexandr P.; Ten Eyck, Lynn F. (2021). «From structure to the dynamic regulation of a molecular switch: A journey over 3 decades». The Journal of Biological Chemistry 296: 100746. ISSN 0021-9258. PMC 8144671. PMID 33957122. doi:10.1016/j.jbc.2021.100746. Consultado el 14 de octubre de 2023.
- ↑ Huang, He; Zhao, Ruijun; Dickson, Bradley M.; Skeel, Robert D.; Post, Carol Beth (2012). «αC Helix as a Switch in the Conformational Transition of Src/CDK-like Kinase Domains». The journal of physical chemistry. B 116 (15): 4465-4475. ISSN 1520-6106. PMC 3630996. PMID 22448785. doi:10.1021/jp301628r. Consultado el 14 de octubre de 2023.
- ↑ Xing, Weirong; Liu, Jeff; Cheng, Shaohong; Vogel, Peter; Mohan, Subburaman; Brommage, Robert (2013). «Targeted disruption of leucine-rich repeat kinase 1 but not leucine-rich repeat kinase 2 in mice causes severe osteopetrosis». Journal of Bone and Mineral Research: The Official Journal of the American Society for Bone and Mineral Research 28 (9): 1962-1974. ISSN 1523-4681. PMC 9528686. PMID 23526378. doi:10.1002/jbmr.1935. Consultado el 22 de octubre de 2023.
- ↑ Shen, Sandi; Si, Mingjue; Zeng, Canjun; Liu, Elaine K.; Chen, Yian; Vacher, Jean; et al. (2023). «Leucine Repeat Rich Kinase 1 Controls Osteoclast Activity by Managing Lysosomal Trafficking and Secretion». Biology 12 (4): 511. ISSN 2079-7737. PMID 37106712. doi:10.3390/biology12040511. Consultado el 22 de octubre de 2023.
- ↑ Guo, Long; Girisha, Katta M; Iida, Aritoshi; Hebbar, Malavika; Shukla, Anju; Shah, Hitesh et al. (2017). «Identification of a novel LRRK1 mutation in a family with osteosclerotic metaphyseal dysplasia». Journal of Human Genetics 62: 437-441. Consultado el 22 de octubre de 2023.
- ↑ Morimoto, Keiko; Baba, Yoshihiro; Shinohara, Hisaaki; Kang, Sujin; Nojima, Satoshi; Kimura, Tetsuya; et al. (2016). «LRRK1 is critical in the regulation of B-cell responses and CARMA1-dependent NF-κB activation». Scientific Reports 6: 25738. ISSN 2045-2322. PMC 4863158. PMID 27166870. doi:10.1038/srep25738. Consultado el 22 de octubre de 2023.
