Proteínas del reloj circadiano de los mamíferos
Las células utilizan una colección compleja de proteínas que se sintetizan y degradan rítmicamente cada día como base del reloj circadiano. Los «osciladores circadianos» son redes de circuitos de retroalimentación bioquímica que generan ritmos de 24 horas en organismos, desde bacterias hasta animales. Estos ritmos periódicos son el resultado de una interacción compleja entre los componentes del reloj que son específicos del organismo, pero comparten mecanismos moleculares entre reinos.
En las células de los mamíferos, la oscilación de 24 horas de los niveles de estas proteínas está controlada por una serie de circuitos de retroalimentación interconectados, donde los niveles de las proteínas regulan con precisión su propia producción.
Las proteínas del reloj circadiano regulan funciones que son críticas como: los niveles de hormonas, el sueño, la temperatura corporal, la presión sanguínea, el metabolismo, el comportamiento, y el nivel de alerta (arousal).[1][2]
Descubrimiento[editar]
El descubrimiento de este sistema circadiano ganó el Premio Nobel en Fisiología y Medicina en 2017 después de que Jeffrey Hall, Michael Rosbash y Michael Young dieran con los mecanismos moleculares que controlan los ritmos del reloj.
El descubrimiento se basa en experimentos realizados en los años ochenta con una mosca del vinagre. Posteriormente se demostró que se trata de un mecanismo biológico muy antiguo, (conservado) a lo largo de la evolución, a medida que se han sucedido las especies.
Hall y Rosbash identificaron en 1984 un gen llamado period, que se encargaba de regular los ritmos circadianos de las moscas. Este también fue identificado de manera independiente por Young. Más tarde, Hall y Rosbash quienes aislaron la proteína PER, que está producida por el gen period. Para explicar cómo esta proteína sincronizaba el ritmo biológico de las moscas propusieron la hipótesis de que la proteína PER inhibe el gen period. De este modo, el gen funcionaría de manera cíclica porque, al activarse, se inhibiría (ya que produciría la proteína PER). Y, al inhibirse, volvería a activarse (ya que el nivel de la proteína PER bajaría). Hall y Rosbash además descubrieron que esta proteína se acumulaba en el núcleo celular durante la noche siguiendo un ciclo diario, pero se sintetizaba en el citoplasma.[3]
Michael Young, en 1994 halló un segundo gen, llamado timeless que produce la proteína TIM. Esta se une a la PER y cuando están juntos pueden entrar al núcleo celular e inhibir el gen period. En los años siguientes, se identificaron más proteínas involucradas en la regulación de los ritmos biológicos.[4]
Localización[editar]
La rotación de la Tierra conduce a ciclos día/noche e impulsa nuestros ritmos circadianos diarios hasta los procesos de microescala a nivel celular. En los mamíferos, la principal señal de tiempo externa (zeitgeber) es el ciclo de luz/oscuridad que transduce señales a través del tracto retinohipotalámico hasta el núcleo supraquiasmático (NSQ) o "reloj maestro" en el cerebro. El NSQ se comunica con el resto del cuerpo para coordinar el ritmo circadiano en cada tejido, da como resultado la regulación molecular de cada reloj celular, lo que permite a los organismos anticiparse y adaptarse a su entorno cambiante.
El NSQ comprende una red neuronal compleja con múltiples conexiones neuronales a través del cerebro y transmite señales a los sistemas nervioso autónomo y endocrino. La influencia del NSQ en el sistema nervioso autónomo altera la sensibilidad de los tejidos periféricos a las hormonas. En consecuencia, un mismo estímulo hormonal puede inducir diferentes respuestas dependiendo de la hora del día. La importancia del NSQ como marcapasos principal se hace evidente cuando el trasplante de un NSQ en funcionamiento restablece el ritmo circadiano en ratones genéticamente arrítmicos; sin embargo, otras señales extracelulares pueden sincronizar de forma independiente los tejidos periféricos.[5]
Estructura[editar]
En los mamíferos, un bucle de retroalimentación de la transcripción genética conforma el reloj circadiano. Se divide en reloj central, que consiste en un TTFL (transcription/translation feedback loop), y reloj periférico.
Las transcripción de las proteínas del reloj central (CLOCK, ARNTL/BMAL1, ARNTL/BMAL2, PER1, PER2, PER3, CRY1, CRY2 y NPAS2) son esenciales para la creación del ritmo circadiano, mientras que sus modificaciones post-transcripcionales funcionan como marcador del periodo (tau: periodo de un ciclo completo circadiano) de estos ritmos.

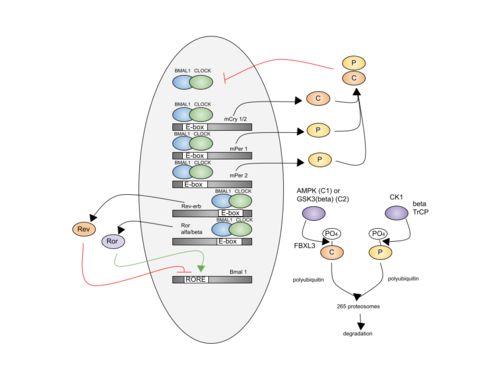
Las proteínas del reloj circadiano son codificadas por varios genes, los más importantes son el gen Clock y el gen Bmal. La proteína CLOCK se encuentra en el núcleo, en el citoplasma y en complejos reguladores de la transcripción. Es codificada por el gen Clock (de sus siglas en inglés "Circadian Locomotor Output Cycles Kaput"), es un factor de transcripción que afecta la duración y la persistencia de los ciclos circadianos. La proteína CLOCK funciona como acetiltransferasa, lo que le permite acerilar proteínas entre las que se encuentran las histonas y la proteína BMAL1/ARTNL, lo que le permite hacer cambios en la cromatina. Esta función acetiltransferasa es importante en el control circadiano de procesos metabólicos. Esta proteína se divide en 3 dominios, dos dominios PAS, presente en muchas proteínas de señalización donde son sensores de señal, y un dominio en motivo hélice-bucle-hélice, típico de los factores de transcripción. Este dominio PAS se ha encontrado en varias proteínas con un papel importante en el desarrollo y la adaptación al medio y que suele ir acompañado del dominio bHLH (hélice-bucle-hélice básica) para funcionar como factor de transcripción heterodímero.[6] Las proteínas Bmal1/ARNTL (Aryl hydrocarbon receptor nuclear translocator-like protein 1), que se encuentra en el citoplasma y en el núcleo, y Bmal 2/ARTNL2, codificadas por el gen Bmal (receptor nuclear translocador de aril hidrocarburos en cerebro y músculo), son factores de transcripción implicados en el reloj circadiano.
BMAL-1, que también contiene un dominio PAS, y CLOCK, se unen formando el complejo BMAL1-CLOCK,[7][8] un heterodímero capaz de dimerizar in vivo y activar la transcripción de los genes Period y Timeless mediante su unión a las secuencias E-box (5’-CACGTG-3’) de sus promotores, que contiene un residuo Ala además de la secuencia habitual de la E-box; aunque también se ha demostrado que reconocía otras secuencias E-box como 5’-AACGTGA-3’ y 5’-CATGTGA-3’. La proteína CLOCK se une a la parte 5’-CAC-3’, mientras que la proteína ARNTL/BMAL1 se une a la secuencia 5’-GTGA-3’.[9] Además, regula los genes del criptocromo (como Cry1 y Cry2) y los genes Period (como Per1, Per2 y Per3) en mamíferos, que a su vez regulan este complejo. Estos factores de transcripción tienen una estructura típica de las proteínas con esta función: el motivo hélice-bucle-hélice.
En un estudio[10] sobre la expresión de los genes del reloj circadiano en 12 órganos en ratones se ha demostrado que, en ratones, casi la mitad (43%) de los genes oscilan con un ritmo circadiano en al menos una parte del organismo, casi siempre de una forma específica para cada órgano. En la mayoría de los casos, la expresión de estas proteínas circadianas era máxima en las llamadas “rush hours” transcripcionales, que corresponden al anochecer y al amanecer. En esta experiencia también se demostraba que los genes circadianos solían estar más agrupados en el genoma, eran más largos y tenían más empalmes (spliceforms) que los genes que no oscilan. También se podía ver que, de los fármacos más utilizados según la OMS, una gran parte tenía como diana productos del ciclo circadiano, y que, por lo tanto, la hora de la toma de estos medicamentos podría afectar su rendimiento. La expresión de los genes circadianos parece ser específica a cada órgano, con poca o ninguna coordinación entre sus genes expresados. En la mayoría de los casos los picos de expresión genética se producen al amanecer y al anochecer. Se puede ver que los genes centrales del reloj circadiano suelen estar sincronizados en todos los órganos; y que los genes con funciones “opuestas” solían tener fases opuestas (ex: activadores vs. inhibidores) El órgano en ratones con más expresión de genes circadianos es, según el experimento, es el hígado, mientras que las tres zonas del cerebro (cerebelo, tronco encefálico e hipotálamo) tenían la menor cantidad de estos genes, aunque la difícil toma de muestras de cerebro hace no segura está segura afirmación. Se puede ver también que solo 10 genes de ratón oscilaban en todos los órganos (Arntl, Dbp, Nr1d1, Nr1d2, Per1, Per2, Per3, Usp2, Tsc22d3 y Tspan4), aunque el estudio de este tipo de mecanismos no es lo suficientemente avanzado para rechazar la presencia de otros genes de este tipo.
Funcionamiento[editar]
El mecanismo del reloj molecular en los mamíferos es un bucle de retroalimentación transcripcional que involucra al menos diez genes. Los genes CLOCK y BMAL codifican bHLH-PAS, proteínas que forman la parte positiva del bucle de retroalimentación. El heterodímero CLOCK:BMAL1 inicia la transcripción uniéndose a elementos específicos del ADN en secuencias E-box (Enhancer box), concretamente en las cajas E (5'-CACGTG-3') y E' (5'-CACGTT-3'). Este conjunto de genes activados incluye proteínas de la parte negativa del bucle de retroalimentación, como las de los genes PER (PER1 y PER2) y CRY (CRY1 y CRY2).[11][12][13]
Las proteínas PER y CRY se dimerizan e inhiben la actividad transcripcional CLOCK:BMAL1, permitiendo que el ciclo se repita desde un nivel bajo de actividad transcripcional.[14][15] Para esta actividad transcripcional cíclica la cromatina se remodela. Esto se logra mediante una combinación de proteínas modificadoras de histonas ubicuas y específicas del reloj.
Se requiere la degradación de las proteínas PER y CRY para terminar la fase de represión de la actividad y reiniciar un nuevo ciclo de transcripción. La tasa estabilidad/degradación de las proteínas PER y CRY es clave para establecer el período del reloj. La caseína quinasa CK1δ fosforila las proteínas PER para la ubicuitinación por βTrCP y la degradación por la proteasoma 26S.[16][17] Las proteínas CRY también se fosforilan y FBXL3 las poliubiquitina.
Los niveles de las proteínas de la parte negativa del bucle, sobre todo las PER, determinan la fase del reloj.[18] Por la noche, cuando los niveles de las proteínas PER son bajos, la administración aguda de luz provoca una inducción de la transcripción PER1 y PER2.[19][20] Con la exposición a la luz en las primeras horas de la noche se observan retrasos en la fase de comportamiento, lo que corresponde a aumentos de las proteínas PER1 y PER2 inducidos por la luz observados en el NSQ.[21] En la segunda mitad de la noche, solo los niveles de PER1 aumentan con la exposición a la luz, lo que corresponde a las fases de la noche en las que se producen avances de fase inducidos por la luz.[21] Los retrasos en el comportamiento en presencia de luz durante el principio de la noche y los avances en la noche o en el alba son suficientes para apoyar el arrastre de un animal hacia un ciclo de luz-oscuridad.
Los dímeros CLOCK:BMAL1 también inician la transcripción de un segundo ciclo de retroalimentación que actúa en coordinación con el ciclo ya descrito. Esto implica la transcripción mediada por E-box de los genes de Rev-Erbα/β y RORα/β (RAR-related orphan receptor). Las proteínas Rev-Erb y ROR luego compiten por los sitios de unión del RORE (Retinoic acid-related Orphan receptor Response Element), donde las proteínas ROR activan la transcripción de BMAL1 y las proteínas Rev-Erb la inhiben.[22][23] Un conjunto separado de genes PAR bZIP que contienen elementos D-box (Destruction box) en sus promotores constituyen otro bucle transcripcional potencial. Estos incluyen genes de la familia HLF, DBP, TEF y Nfil3.[24][25][26][27]
Si considerásemos solo la tasa transcripción/traducción y el bucle de transcripción E-box descrito solo para los genes PER/CRY, el ciclo completo debería tomar significativamente menos de un día o incluso pocas horas. Para resolver este problema, se ha planteado que los tres elementos que proporcionan el retraso necesario para completar el ciclo cerca de las 24 horas son: E-box por la mañana, D-box durante el día y elementos RORE por la noche.[28] Aunque no se requieran genes en estos bucles accesorios de D-box para la función del reloj, pueden servir para hacer que las oscilaciones del núcleo sean más estables y agregar precisión al período del reloj.[22][29]
Otras funciones[editar]
El reloj circadiano central y los relojes periféricos que residen en los tejidos metabólicamente activos están bien coordinados para conferir una homeostasis metabólica coherente.[30]
Regulación del metabolismo lipídico[editar]
El reloj circadiano (CC en inglés) regula las vías de metabolismo de lípidos en todos los órganos metabólicos principales. En el hígado, todos los miembros de la familia proteica PPAR están regulados diurnamente.[31] Se ha observado experimentalmente, que PPARα y PGC-1α se ciclan juntos y promueven la β-oxidación de ácidos grasos en las mitocondrias al comienzo de la noche en ratones, coincidiendo con el inicio de la búsqueda de alimento.[32]
El reloj circadiano también es necesario para la expresión circadiana de las enzimas limitantes de la velocidad para la síntesis de colesterol y ácidos biliares, tales como HMG-CoA reductasa y colesterol 7α-hidroxilasa. El sistema circadiano asegura que la síntesis de ácidos biliares sea más alta durante el periodo en que se consume la mayor proporción de alimentos.
El reloj circadiano tiene importancia en la regulación de la tasa de lipólisis en el tejido adiposo blanco, donde se almacenan grandes cantidades de triglicéridos. En los mamíferos, los genes de la lipasa de los triglicéridos adiposos (ATGL) y la lipasa sensible a hormonas (HSL) son activados directamente por el complejo transcripcional CLOCK:BMAL1. Esto permite que el reloj central module la velocidad de la hidrólisis de lípidos a lo largo del ciclo circadiano.[33]
Patologías Asociadas[editar]
Cáncer[editar]
Las células tumorales se caracterizan por una proliferación celular incontrolada que da como resultado un crecimiento tisular anormal y acelerado. Por el contrario, las células "sanas" a menudo proliferan con una tasa de división de 24 h. Esto se debe al control directo de los puntos de control del ciclo celular por parte de la maquinaria del reloj circadiano intracelular.
En cualquier tipo de célula, el 5-20% del transcriptoma está bajo control circadiano. Esta es la base para el control circadiano de los principales procesos fisiológicos, incluidas las funciones inmunitarias y la proliferación celular.
La desalineación entre el tiempo externo e interno y la alteración circadiana, como durante el trabajo por turnos, se ha asociado con un mayor riesgo de cáncer y promueve el crecimiento tumoral. Además, la alteración del reloj circadiano debido a mutaciones de genes de reloj único, como Per2 o Bmal1, acelera el crecimiento tumoral o todo el proceso de carcinogénesis. Esto se debe supuestamente a un aumento en la tasa de proliferación tras la alteración del ritmo circadiano, porque los genes supresores de tumores y los genes clave del ciclo celular están bajo el control del reloj en el contexto de un acoplamiento bidireccional reloj-ciclo celular.
El pronóstico y la supervivencia del cáncer se han asociado con el nivel de alteración circadiana en los tejidos tumorales de los pacientes. Se ha demostrado que la mejora de los ritmos circadianos en el tumor ralentiza la progresión del ciclo celular y reduce fuertemente la proliferación y el crecimiento tumoral.[34]
Neurodegeneración[editar]
El envejecimiento cerebral se asocia con una disminución de la producción del reloj circadiano y una disminución de la expresión de las proteínas del reloj central, que regulan muchos aspectos de la bioquímica y el metabolismo celular. La deleción de los activadores de la transcripción del reloj circadiano, el receptor de hidrocarburos de arilo, el translocador nuclear (Bmal1) solo, o los ciclos de producción locomotora circadiana kaput (Clock) en combinación con la proteína del dominio PAS neuronal 2 (Npas2), inducen una astrogliosis grave dependiente de la edad en el paciente.
En los tejidos periféricos, los genes reloj sirven como reguladores críticos del metabolismo celular y la homeostasis redox, y se han implicado en el proceso de envejecimiento.[35][36] Los ratones con deleción dirigida de Bmal1 muestran pérdida de los ritmos circadianos conductuales y fisiológicos y desarrollan un mayor estrés oxidativo sistémico y signos de envejecimiento acelerado.[37][38] Por el contrario, el envejecimiento se asocia con una expresión disminuida de los genes del reloj de extremidades positivas en el cerebro del ratón. La oscilación circadiana alterada y la lesión oxidativa se asocian con el envejecimiento cerebral y las condiciones neurodegenerativas relacionadas con la edad en los seres humanos, lo que sugiere un posible vínculo entre la disfunción del reloj circadiano, el estrés oxidativo y la neurodegeneración relacionada con la edad.[39] Sin embargo, se desconoce si los genes del reloj central juegan algún papel en el mantenimiento de la salud neuronal o si estos genes influyen en la neurodegeneración.
BMAL1 se ha implicado en la función astrocítica y del hipocampo,[40][41] este en un complejo con CLOCK o NPAS2 regula la homeostasis redox cerebral y conecta la función alterada del gen del reloj con la neurodegeneración. La deleción de Bmal1 causa neuropatología dependiente de la edad y degeneración sináptica. Por lo tanto, conduce a una astrocitosis generalizada, así como a una degeneración discreta de las terminales axonales presinápticas.[42]
Referencias[editar]
- ↑ Más, Paloma (2019). «Cellular and molecular mechanisms of circadian clock function in Arabidopsis thaliana». CSIC ORCID.
- ↑ «Ritmos circadianos». NIGMS National Institute of General Medical Sciences. 2022.
- ↑ «The 2017 Nobel Prize in Physiology or Medicine - Press Release». 2017.
- ↑ «El reloj biológico del cuerpo humano gana el Nobel de Medicina». La Vanguardia. 2 de octubre de 2017. Consultado el 11 de noviembre de 2021.
- ↑ Borrmann, Helene; McKeating, Jane A.; Zhuang, Xiaodong (2021-2). «The Circadian Clock and Viral Infections». Journal of Biological Rhythms 36 (1): 9-22. ISSN 0748-7304. PMC 7924106. PMID 33161818. doi:10.1177/0748730420967768. Consultado el 8 de noviembre de 2021.
- ↑ Huang, Z. J.; Edery, I.; Rosbash, M. (15 de julio de 1993). «PAS is a dimerization domain common to Drosophila period and several transcription factors». Nature 364 (6434): 259-262. ISSN 0028-0836. PMID 8391649. doi:10.1038/364259a0. Consultado el 10 de noviembre de 2021.
- ↑ «CLOCK - Circadian locomoter output cycles protein kaput - Homo sapiens (Human) - CLOCK gene & protein». Uniprot.org (en inglés). Consultado el 10 de noviembre de 2021.
- ↑ Turek, Fred W.; Joshu, Corinne; Kohsaka, Akira; Lin, Emily; Ivanova, Ganka; McDearmon, Erin; Laposky, Aaron; Losee-Olson, Sue et al. (13 de mayo de 2005). «Obesity and metabolic syndrome in circadian Clock mutant mice». Science (New York, N.Y.) 308 (5724): 1043-1045. ISSN 1095-9203. PMC 3764501. PMID 15845877. doi:10.1126/science.1108750. Consultado el 10 de noviembre de 2021.
- ↑ «Intermolecular recognition revealed by the complex structure of human CLOCK-BMAL1 basic helix-loop-helix domains with E-box DNA.». www.uniprot.org (en inglés). Consultado el 10 de noviembre de 2021.
- ↑ Zhang, Ray; Lahens, Nicholas F.; Ballance, Heather I.; Hughes, Michael E.; Hogenesch, John B. (11 de noviembre de 2014). «A circadian gene expression atlas in mammals: Implications for biology and medicine». Proceedings of the National Academy of Sciences (en inglés) 111 (45): 16219-16224. ISSN 0027-8424. PMID 25349387. doi:10.1073/pnas.1408886111. Consultado el 10 de noviembre de 2021.
- ↑ Gekakis, N.; Staknis, D.; Nguyen, H. B.; Davis, F.C.; Wilsbacher, L.D.; King, D.P.; Takahashi, J. S.; Weitz, C.J. (5 de junio de 1998). «Role of the CLOCK protein in the mammalian circadian mechanism». Science (New York, N.Y.) 280 (5369): 1564-1569. ISSN 0036-8075. PMID 9616112. doi:10.1126/science.280.5369.1564. Consultado el 8 de noviembre de 2021.
- ↑ Hogenesch, J. B.; Gu, Y. Z.; Jain, S.; Bradfield, C. A. (12 de mayo de 1998). «The basic-helix-loop-helix-PAS orphan MOP3 forms transcriptionally active complexes with circadian and hypoxia factors». Proceedings of the National Academy of Sciences of the United States of America 95 (10): 5474-5479. ISSN 0027-8424. PMID 9576906. doi:10.1073/pnas.95.10.5474. Consultado el 9 de noviembre de 2021.
- ↑ Kume, K.; Zylka, M.J.; Sriram, S.; Shearman, L.P.; Weaver, D. R.; Jin, X.; Maywood, E.S.; Hastings, M. H. et al. (23 de julio de 1999). «mCRY1 and mCRY2 are essential components of the negative limb of the circadian clock feedback loop». Cell 98 (2): 193-205. ISSN 0092-8674. PMID 10428031. doi:10.1016/s0092-8674(00)81014-4. Consultado el 9 de noviembre de 2021.
- ↑ Griffin, E. A.; Staknis, D.; Weitz, C. J. (22 de octubre de 1999). «Light-independent role of CRY1 and CRY2 in the mammalian circadian clock». Science (New York, N.Y.) 286 (5440): 768-771. ISSN 0036-8075. PMID 10531061. doi:10.1126/science.286.5440.768. Consultado el 9 de noviembre de 2021.
- ↑ Sato, Trey K.; Yamada, Rikuhiro G.; Ukai, Hideki; Baggs, Julie E.; Miraglia, Loren J.; Kobayashi, Tetsuya J.; Welsh, David K.; Kay, Steve A. et al. (2006-03). «Feedback repression is required for mammalian circadian clock function». Nature Genetics 38 (3): 312-319. ISSN 1061-4036. PMC 1994933. PMID 16474406. doi:10.1038/ng1745. Consultado el 9 de noviembre de 2021.
- ↑ Camacho, F.; Cilio, M.; Guo, Y.; Virshup, D. M.; Patel, K.; Khorkova, O.; Styren, S.; Morse, B. et al. (2 de febrero de 2001). «Human casein kinase Idelta phosphorylation of human circadian clock proteins period 1 and 2». FEBS letters 489 (2-3): 159-165. ISSN 0014-5793. PMID 11165242. doi:10.1016/s0014-5793(00)02434-0. Consultado el 9 de noviembre de 2021.
- ↑ Vanselow, Katja; Vanselow, Jens T.; Westermark, Pål O.; Reischl, Silke; Maier, Bert; Korte, Thomas; Herrmann, Andreas; Herzel, Hanspeter et al. (1 de octubre de 2006). «Differential effects of PER2 phosphorylation: molecular basis for the human familial advanced sleep phase syndrome (FASPS)». Genes & Development 20 (19): 2660-2672. ISSN 0890-9369. PMC 1578693. PMID 16983144. doi:10.1101/gad.397006. Consultado el 9 de noviembre de 2021.
- ↑ Edery, I.; Rutila, J. E.; Rosbash, M. (14 de enero de 1994). «Phase shifting of the circadian clock by induction of the Drosophila period protein». Science (New York, N.Y.) 263 (5144): 237-240. ISSN 0036-8075. PMID 8284676. doi:10.1126/science.8284676. Consultado el 9 de noviembre de 2021.
- ↑ Albrecht, U.; Sun, Z. S.; Eichele, G.; Lee, C. C. (26 de diciembre de 1997). «A differential response of two putative mammalian circadian regulators, mper1 and mper2, to light». Cell 91 (7): 1055-1064. ISSN 0092-8674. PMID 9428527. doi:10.1016/s0092-8674(00)80495-x. Consultado el 9 de noviembre de 2021.
- ↑ Shigeyoshi, Y.; Taguchi, K.; Yamamoto, S.; Takekida, S.; Yan, L.; Tei, H.; Moriya, T.; Shibata, S. et al. (26 de diciembre de 1997). «Light-induced resetting of a mammalian circadian clock is associated with rapid induction of the mPer1 transcript». Cell 91 (7): 1043-1053. ISSN 0092-8674. PMID 9428526. doi:10.1016/s0092-8674(00)80494-8. Consultado el 9 de noviembre de 2021.
- ↑ a b Yan, Lily; Silver, Rae (2004-02). «Resetting the brain clock: time course and localization of mPER1 and mPER2 protein expression in suprachiasmatic nuclei during phase shifts». The European Journal of Neuroscience 19 (4): 1105-1109. ISSN 0953-816X. PMC 3271804. PMID 15009158. doi:10.1111/j.1460-9568.2004.03189.x. Consultado el 9 de noviembre de 2021.
- ↑ a b Preitner, Nicolas; Damiola, Francesca; Lopez-Molina, Luis; Zakany, Joszef; Duboule, Denis; Albrecht, Urs; Schibler, Ueli (26 de julio de 2002). «The orphan nuclear receptor REV-ERBalpha controls circadian transcription within the positive limb of the mammalian circadian oscillator». Cell 110 (2): 251-260. ISSN 0092-8674. PMID 12150932. doi:10.1016/s0092-8674(02)00825-5. Consultado el 9 de noviembre de 2021.
- ↑ Guillaumond, Fabienne; Dardente, Hugues; Giguère, Vincent; Cermakian, Nicolas (2005-10). «Differential control of Bmal1 circadian transcription by REV-ERB and ROR nuclear receptors». Journal of Biological Rhythms 20 (5): 391-403. ISSN 0748-7304. PMID 16267379. doi:10.1177/0748730405277232. Consultado el 9 de noviembre de 2021.
- ↑ Falvey, E.; Fleury-Olela, F.; Schibler, U. (1 de septiembre de 1995). «The rat hepatic leukemia factor (HLF) gene encodes two transcriptional activators with distinct circadian rhythms, tissue distributions and target preferences». The EMBO journal 14 (17): 4307-4317. ISSN 0261-4189. PMID 7556072. Consultado el 9 de noviembre de 2021.
- ↑ Lopez-Molina, L.; Conquet, F.; Dubois-Dauphin, M.; Schibler, U. (17 de noviembre de 1997). «The DBP gene is expressed according to a circadian rhythm in the suprachiasmatic nucleus and influences circadian behavior». The EMBO journal 16 (22): 6762-6771. ISSN 0261-4189. PMC 1170280. PMID 9362490. doi:10.1093/emboj/16.22.6762. Consultado el 9 de noviembre de 2021.
- ↑ Fonjallaz, P.; Ossipow, V.; Wanner, G.; Schibler, U. (15 de enero de 1996). «The two PAR leucine zipper proteins, TEF and DBP, display similar circadian and tissue-specific expression, but have different target promoter preferences». The EMBO journal 15 (2): 351-362. ISSN 0261-4189. PMID 8617210. Consultado el 9 de noviembre de 2021.
- ↑ Mitsui, S.; Yamaguchi, S.; Matsuo, T.; Ishida, Y.; Okamura, H. (15 de abril de 2001). «Antagonistic role of E4BP4 and PAR proteins in the circadian oscillatory mechanism». Genes & Development 15 (8): 995-1006. ISSN 0890-9369. PMID 11316793. doi:10.1101/gad.873501. Consultado el 9 de noviembre de 2021.
- ↑ Ukai-Tadenuma, Maki; Yamada, Rikuhiro G.; Xu, Haiyan; Ripperger, Jürgen A.; Liu, Andrew C.; Ueda, Hiroki R. (21 de enero de 2011). «Delay in feedback repression by cryptochrome 1 is required for circadian clock function». Cell 144 (2): 268-281. ISSN 1097-4172. PMID 21236481. doi:10.1016/j.cell.2010.12.019. Consultado el 9 de noviembre de 2021.
- ↑ Liu, Andrew C.; Tran, Hien G.; Zhang, Eric E.; Priest, Aaron A.; Welsh, David K.; Kay, Steve A. (29 de febrero de 2008). «Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms». PLoS genetics 4 (2): e1000023. ISSN 1553-7404. PMC 2265523. PMID 18454201. doi:10.1371/journal.pgen.1000023. Consultado el 9 de noviembre de 2021.
- ↑ Swetha Gopalakrishnan, Nisha N. Kannan (2021). «Only time will tell: The interplay between circadian clock and metabolism». Chronobiol Int. 38 (2): 149-167. PMC 7611229.
- ↑ Yang, Xiaoyong; Downes, Michael; Yu, Ruth T.; Bookout, Angie L.; He, Weimin; Straume, Marty; Mangelsdorf, David J.; Evans, Ronald M. (25 de agosto de 2006). «Nuclear receptor expression links the circadian clock to metabolism». Cell 126 (4): 801-810. ISSN 0092-8674. PMID 16923398. doi:10.1016/j.cell.2006.06.050. Consultado el 11 de noviembre de 2021.
- ↑ Li, Min-Dian; Li, Chao-Min; Wang, Zhong (2012-09). «The role of circadian clocks in metabolic disease». The Yale Journal of Biology and Medicine 85 (3): 387-401. ISSN 1551-4056. PMC 3447202. PMID 23012586. Consultado el 11 de noviembre de 2021.
- ↑ Shostak, Anton; Meyer-Kovac, Judit; Oster, Henrik (2013-07). «Circadian regulation of lipid mobilization in white adipose tissues». Diabetes 62 (7): 2195-2203. ISSN 1939-327X. PMC 3712056. PMID 23434933. doi:10.2337/db12-1449. Consultado el 11 de noviembre de 2021.
- ↑ Kiessling, Silke; Beaulieu-Laroche, Lou; Blum, Ian D.; Landgraf, Dominic; Welsh, David K.; Storch, Kai-Florian; Labrecque, Nathalie; Cermakian, Nicolas (14 de febrero de 2017). «Enhancing circadian clock function in cancer cells inhibits tumor growth». BMC Biology 15: 13. ISSN 1741-7007. PMC 5310078. PMID 28196531. doi:10.1186/s12915-017-0349-7. Consultado el 8 de noviembre de 2021.
- ↑ Bass, Joseph; Takahashi, Joseph S. (3 de diciembre de 2010). «Circadian Integration of Metabolism and Energetics». Science (en inglés) 330 (6009): 1349-1354. ISSN 0036-8075. doi:10.1126/science.1195027. Consultado el 10 de noviembre de 2021.
- ↑ Rutter, Jared; Reick, Martin; Wu, Leeju C.; McKnight, Steven L. (20 de julio de 2001). «Regulation of Clock and NPAS2 DNA Binding by the Redox State of NAD Cofactors». Science (en inglés) 293 (5529): 510-514. ISSN 0036-8075. doi:10.1126/science.1060698. Consultado el 10 de noviembre de 2021.
- ↑ Kondratov, R. V. (15 de julio de 2006). «Early aging and age-related pathologies in mice deficient in BMAL1, the core componentof the circadian clock». Genes & Development (en inglés) 20 (14): 1868-1873. ISSN 0890-9369. doi:10.1101/gad.1432206. Consultado el 10 de noviembre de 2021.
- ↑ Bunger, Maureen K.; Wilsbacher, Lisa D.; Moran, Susan M.; Clendenin, Cynthia; Radcliffe, Laurel A.; Hogenesch, John B.; Simon, M.Celeste; Takahashi, Joseph S. et al. (2000-12). «Mop3 Is an Essential Component of the Master Circadian Pacemaker in Mammals». Cell (en inglés) 103 (7): 1009-1017. doi:10.1016/S0092-8674(00)00205-1. Consultado el 10 de noviembre de 2021.
- ↑ Wyse, Cathy A.; Coogan, Andrew N. (2010-06). «Impact of aging on diurnal expression patterns of CLOCK and BMAL1 in the mouse brain». Brain Research (en inglés) 1337: 21-31. doi:10.1016/j.brainres.2010.03.113. Consultado el 10 de noviembre de 2021.
- ↑ Rath, Martin F.; Rohde, Kristian; Fahrenkrug, Jan; Møller, Morten (2013-03). «Circadian clock components in the rat neocortex: daily dynamics, localization and regulation». Brain Structure and Function (en inglés) 218 (2): 551-562. ISSN 1863-2653. doi:10.1007/s00429-012-0415-4. Consultado el 10 de noviembre de 2021.
- ↑ Valnegri, Pamela; Khelfaoui, Malik; Dorseuil, Olivier; Bassani, Silvia; Lagneaux, Celine; Gianfelice, Antonella; Benfante, Roberta; Chelly, Jamel et al. (28 de agosto de 2011). «A circadian clock in hippocampus is regulated by interaction between oligophrenin-1 and Rev-erbα». Nature Neuroscience 14 (10): 1293-1301. ISSN 1097-6256. doi:10.1038/nn.2911. Consultado el 10 de noviembre de 2021.
- ↑ Musiek, Erik S.; Lim, Miranda M.; Yang, Guangrui; Bauer, Adam Q.; Qi, Laura; Lee, Yool; Roh, Jee Hoon; Ortiz-Gonzalez, Xilma et al. (2 de diciembre de 2013). «Circadian clock proteins regulate neuronal redox homeostasis and neurodegeneration». The Journal of Clinical Investigation 123 (12): 5389-5400. ISSN 0021-9738. PMC 3859381. PMID 24270424. doi:10.1172/JCI70317. Consultado el 10 de noviembre de 2021.
Enlaces externos[editar]
Página sobre Ritmos circadianos del NIH (en español).