Unión de Holliday

Una unión de Holliday (del inglés Holliday junction, HJ) es una estructura de ácido nucleico ramificada en la que están unidos cuatro dominios de doble cadena de ácido nucleico. En biología, las uniones de Holliday funcionan como intermediarios en la recombinación genética; estas uniones o cruces (junctions) tienen generalmente una secuencia simétrica, por lo que son móviles. Las uniones de Holliday inmóviles fueron creadas artificialmente por los científicos inicialmente para estudiar sus estructuras como modelo de las uniones de Holliday, pero posteriormente encontraron un uso como materiales de construcción estructurales básicos en nanotecnología del ADN. La estructura recibe su nombre por Robin Holliday, que propuso su existencia en 1964.
Estructura[editar]
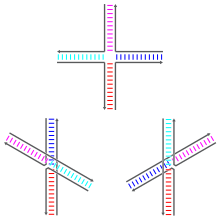
Las uniones de Holliday pueden existir en forma de varios isómeros conformacionales con diferentes patrones de amontonamiento coaxial (coaxial stacking) entre los cuatro dominios de doble hélice.[1] El amontonamiento coaxial es la tendencia de los extremos romos (es decir, no coherentes) de ácidos nucleicos de unirse uno con otro debido a las interacciones entre los anillos de las bases expuestas. Hay tres posibles conformaciones de amontonamiento: una forma no amontonada (unstacked) y dos amontonadas (stacked). La forma no amontonada domina en ausencia de cationes divalentes como el Mg2+ debido a la repulsión electrostática entre los esqueletos cargados negativamente de las hebras, mientras que en presencia de por lo menos un 0,1 mm de Mg2+ a repulsión electrostática es contrarrestada y predomina la estructura amontonada. En 2000, no se conocía con certeza si el apantallamiento electrostático era el resultado de la unión específica de sitio de cationes a la unión de Holliday, o de la presencia de una colección difusa de iones en solución.[2]
La forma no amontonada es una conformación extendida casi plana y cuadrada. Por otra parte, los confórmeros amontonados tienen dos dominios en doble hélice continuos separados por un ángulo de unos 60° hacia la derecha. Dos de las cuatro fibras permanecen aproximadamente helicoidales, y quedan dentro de los dos dominios en doble hélice, mientras que los otros dos cruzan entre los dos dominios en disposición antiparalela.[2]
Las dos formas amontonadas posibles se diferencian en las identidades que tengan los pares de brazos que están amontonados uno sobre lo otro, y cual domine dependerá en gran medida de las secuencias de bases próximas a la unión. Algunas secuencias dan lugar a un equilibrio entre los dos confórmeros, mientras que otras prefieren claramente un solo confórmero. En concreto, las uniones que contienen la secuencia A-CC que hace de puente en el punto de unión parecen preferir claramente el confórmero que permite que se forme un enlace de hidrógeno entre la segunda citosina y uno de los fosfatos del punto de unión. Aunque la mayoría de los estudios se enfocaron a estudiar las identidades de las cuatro bases próximas a la unión en cada brazo, es evidente que las bases que están más lejos pueden también afectar a las conformaciones amontonadas observadas.[2]
En uniones con secuencias simétricas, el punto de ramificación es móvil y puede migrar en un proceso aleatorio. La velocidad de migración de la rama varía drásticamente con la concentración de iones, y los tiempos para dar un solo paso se incrementan desde 0,3−0,4 milisegundos (ms) sin ningún ion a 270−300 ms con 10 mm de Mg2+. El cambio en la velocidad está correlacionado con la formación de estructuras amontonadas o no amontonadas.[2]
Las uniones de Holliday con una muesca o rotura en una de las hebras, en el punto de unión adoptan una orientación perpendicular, y siempre prefieren el confórmero amontonado que sitúa la muesca en una fibra que sobrecruza en vez de la fibra helicoidal.[2]
Las uniones de Holliday de ARN se conocen en algunos ARNs biológicos funcionales, como el ARN espliceosomal U1 y en el ribocima forquita del virus de la mancha anular del tabaco. Las uniones de Holliday de ARN adoptan una conformación amontonada antiparalela a altas concentraciones de magnesio, una conformación amontonada perpendicular a concentraciones moderadas, y rotan en una conformación amontonada paralela a bajas concentraciones, mientras que incluso pequeñas concentraciones del ion calcio (Ca2+) favorecen el confórmero antiparalelo.[2]
Función biológica[editar]

La unión de Holliday es un intermediario clave en la recombinación homóloga, un proceso biológico que incrementa la diversidad genética al intercambiar genes entre dos cromosomas, y también en eventos de recombinación específica de sitio en los que intervienen integrasas. Además, están implicadas en la reparación de roturas de doble fibra.[2] Adicionalmente, pueden originarse estructuras cruciformes que implican a las uniones de Holliday para aliviar la tensión en la hélice de secuencias simétricas en ADN superenrollado.[3] Aunque las uniones de cuatro brazos también aparecen en moléculas de ARN funcionales, estas generalmente contienen nucleótidos no apareados situados entre los dominios de doble hélice apareados, y no adoptan estrictamente la estructura de Holliday.[2]
Las uniones de Holliday en la recombinación homóloga están entre secuencias idénticas o casi idénticas, lo que lleva a un arreglo simétrico de secuencias alrededor de la unión central. Esto permite que ocurra un proceso de migración de la rama, en el que las fibras se mueven a través del punto de unión.[2] La clivaxe (corte) o la resolución de la unión de Holliday puede ocurrir de dos maneras. La separación del conjunto original de fibras origina dos moléculas que pueden presentar conversión génica pero no sobrecruzamiento cromosómico, mientras que la separación del otro conjunto de dos fibras causa que las moléculas recombinantes resultantes presenten sobrecruzamiento. Todos los productos, independientemente de su separación, son heterodúplex en la región de la migración de la unión de Holliday.[4]
Muchas proteínas pueden reconocer o distorsionar la estructura de la unión de Holiday. Una de esas clases contiene enzimas que resuelven la unión que se encargan de adherir las uniones, a veces de un modo específico de secuencia. Dichas proteínas distorsionan la estructura de la unión de varios modos, a menudo empujando la unión a adoptar una conformación no amontonada (unstacked), rompiendo los pares de bases centrales, y/o cambiando los ángulos entre los cuatro brazos. Otras clases son las proteínas de migración de la rama, que incrementan la velocidad de intercambio en varios órdenes de magnitud, y las recombinasas específicas de sitio.[2] En procariotas, las resoluciones de la unión de Holliday pueden pertenecer la dos familias, integrasas y nucleasas, que son cada una de ellas estructuralmente similares, aunque sus secuencias no están conservadas.[4]
En eucariotas, hay dos modelos principales para explicar como la recombinación homóloga repara las roturas de doble fibra en el ADN, que son la vía de reparación de roturas de doble fibra (DSBR) (a veces llamado modelo de unión de Holliday doble) y la vía de alineamiento (annealing) de fibras dependiente de síntesis (SDSA).[5] En el caso de rotura de doble fibra, el extremo 3' es degradado y el extremo 5' más grande invade la cromátida hermana contigua, formando una burbuja de replicación. A medida que la burbuja se acerca al ADN roto, la fibra antisentido 5' más larga invade de nuevo la fibra sentido de esta porción del ADN, transcribiendo una segunda copia. Cuando termina la replicación, ambas colas son reconectadas para formar dos uniones de Holliday, que son después unidas por proteínas de variadas maneras.[6] Una animación de este proceso puede verse aquí.[7]
Las roturas en el ADN de doble fibra en bacterias son reparadas por la vía RecBCD de recombinación homóloga. Las roturas que ocurren solamente en una de las dos fibras del ADN, y que se conocen como huecos (gaps) de fibra simple, se creen que son reparadas por la vía RecF. Tanto la vía RecBCD como a RecF incluyen una serie de reacciones conocidas como migración de la rama, en la cual las fibras de ADN sencillas se intercambian entre dos moléculas entrecruzadas de ADN dúplex, y también la resolución, en la cual las dos moléculas de ADN que se habían entrecruzado son cortadas y restauradas a su estado normal de doble fibra.[8] La recombinación homóloga ocurre en varios grupos de virus. En virus de ADN como los herpesvirus, la recombinación ocurre por medio de un mecanismo de rotura y reunión como en bacterias y eucariotas.[9] En bacterias, la migración de rama está facilitada lo pones complejo RuvABC o la proteína RecG, que son motores moleculares que usan la energía de la hidrólisis de ATP para mover la unión. La unión debe después ser resuelta en dos dúplex separados, restaurando la configuración parental o una configuración sobrecruzada. La resolución puede ocurrir de manera horizontal o vertical durante la recombinación homóloga, dando lugar a productos parche (están en la misma orientación durante la reparación de las roturas de doble fibra) o a productos de empalme (están en diferentes orientaciones durante la reparación de roturas de doble fibra).[10][11] RuvA y RuvB son proteínas de la migración de la rama, mientras que RuvC es un enzima que resuelve la unión.[2]
Hay también pruebas de recombinación en algunos virus de ARN, específicamente en virus de ARN monocatenario de sentido positivo como los retrovirus, picornavirus, y coronavirus. Hay controversia sobre se la recombinación homóloga ocurre en virus de ARN monocatenario de sentido negativo cómo lo de la gripe.[12]
Uso en nanotecnología del ADN[editar]


La nanotecnología del ADN es el diseño y manufactura de estructuras de ácido nucleico artificiales utilizadas como material de ingeniería para nanotecnología en vez de como portadores de la información genética en las células vivas. En este campo se utilizan estructuras de ADN ramificadas como componentes fundamentales para crear estructuras diseñadas racionalmente más complejas. Las uniones de Holliday son componentes de muchas de estas estructuras de ADN. Como los complejos de uniones de Holliday aislados son demasiado flexibles para ensamblarse en matrices (arrays) ordenadas grandes, los motivos estructurales con muchas uniones de Holliday se utilizan para crear "baldosas" rígidas que pueden después ensamblarse en "matrices" más grandes.[14][15]
El motivo de este tipo más común es el complejo de doble sobrecruzamiento (DX), que contiene dos uniones de Holliday en estrecha cercanía una de la otra, que da lugar a una estructura rígida que puede autoensamblarse en matrices más grandes. La estructura de la molécula DX fuerza a las uniones de Holliday a adoptar una conformación con los dominios en doble hélice situados directamente lado con lado, en contraste con su ángulo preferido de aproximadamente 60°. El complejo puede teóricamente ser diseñado para forzar las uniones a tener una orientación paralela o antiparalela, pero en la práctica las variedades antiparalelas se comportan mejor, y las versiones paralelas raramente si usan.[14][15]
El motivo estructural DX es también la pieza de construcción fundamental del método ADN origami, que se utiliza para hacer estructuras bi o tridimensionles más grandes de forma arbitraria. En lugar de usar baldosas DX individuales, se pliega una hebra de entramado largo sencillo en la forma deseada usando varias fibras grapa cortas. Cuando se ensamblan, la fibra entramado es continua a través de los dominios de doble hélice, mientras que las fibras grapa participan en las uniones de Holliday como fibras de sobrecruzamiento.[16]
Se demostró que hay varios tipos de baldosas que mantienen el ángulo nativo de 60° de las uniones de Holliday. Una de estas matrices usa baldosas que contienen cuatro uniones de Holliday en una disposición en paralelogramo. Esta estructura tenía la ventaja de permitir que el ángulo de la unión sea visualizado directamente por medio de microscopia de fuerza atómica. Además, se utilizaron las baldosas de tres uniones de Holliday en disposición triangular basadas en el principio de la integridad tensional para construir matrices tridimensionales periódicas pensadas para usarlas en cristalografía de rayos X de biomoléculas.[14][15]
Historia[editar]
Robin Holliday propuso esta estructura como parte de su modelo de recombinación homóloga en 1964, basado en su investigación en los organismos Ustilago maydis y Saccharomyces cerevisiae. El modelo proporciona un mecanismo molecular que explicaba tanto la conversión génica como el sobrecruzamiento cromosómico. Holliday se enteró de que la vía propuesta crearía segmentos de ADN heterodúplex con bases mal apareadas entre diferentes versiones de un solo gen, y postuló que la célula debe tener un mecanismo para reparar estas discordancias en el apareamiento de bases, lo cual si descubrió más tarde.[4] Antes del modelo de Holliday, el modelo aceptado implicaba el mecanismo de elección de copia.[17]
En el modelo de Holliday original de recombinación homóloga, las roturas de una sola hebra aparecían en el mismo punto de una hebra de cada ADN parental. Los extremos libres de cada hebra rota migran después a través de la otra hélice de ADN, en la que las hebras invasoras se unen a los extremos libres que encuentren, lo que tiene como resultado a formación de la unión de Holliday. Como cada hebra sobrecruzada se realinea (reanneals) con su fibra compañera original, desplaza la fibra complementaria original hacia delante, haciendo que la unión de Holliday migre, creando los segmentos heterodúplex. Dependiendo de qué hebra se utilizó como modelo para reparar a otra, las cuatro células resultantes de una meiosis podrían acabar con tres copias de un alelo y sólo una del otro, en vez de las dos de cada normales, una propiedad conocida como conversión génica.[4]
El modelo original de Holliday asumía que el ADN heterodúplex estaría presente en ambos cromosomas pero los datos experimentales obtenidos en levaduras refutaron esta idea, lo que llevó a que el modelo fuera puesto al día en 1975 por Matt Meselson y Charley Radding, que introdujeron la idea de la migración de la rama.[17] Posteriores observaciones hechas en la década de 1980 llevaron a proponer mecanismos alternativos para la recombinación como el modelo de rotura de doble fibra (de Jack Szostak, Frank Stahl, y otros) y el modelo de alineamiento (annealing) de hebra sencilla. Un tercer modelo, llamado modelo de alineamiento de fibra dependiente de síntesis, no implica uniones de Holliday.[4]
La primera evidencia experimental de la estructura de las uniones de Holliday procede de estudios de microscopia electrónica hechos al final de la década de 1970, en los que la estructura de cuatro brazos era claramente visible en imágenes de ADN de plásmidos y bacteriófagos. Posteriormente en la década de 1980, se identificaron las enzimas responsables para iniciar la formación de las uniones de Holliday y cómo se unen ellas, aunque en 2004 la identificación de las resoluciones de uniones de Holliday de mamíferos seguía siendo evasiva. En 1983, se construyeron las primeras moléculas de unión de Holliday artificiales a partir de oligonucleótidos sintéticos gracias a los trabajos de Nadrian Seeman, lo que permitió un estudio más directo de sus propiedades físicas. Muchas de los análisis tempranos de las estructuras de unión de Holliday se infirieron de estudios de electroforesis en gel, FRET, y huellas de nucleasas y de radical hidroxilo. En la década de 1990, los métodos de cristalografía y RMN de ácidos nucleicos ya estaban disponibles, y también las herramientas informáticas de modelado molecular.[2][4][18]
Inicialmente, los genetistas asumieron que la unión adoptaría una conformación paralela antes que antiparalela porque esta situaría los dúplex homólogos en alineamiento muy próximo.[2] Pero los análisis químicos hechos en la década de 1980 mostraron que, en realidad, la unión prefería las conformaciones antiparalelas, lo que fue un descubrimiento considerado controvertido, y el propio Robin Holliday dudaba inicialmente de él.[2][4] No obstante, posteriormente, la estructura antiparalela fue ampliamente aceptada debido a datos de cristalografía de rayos X en moléculas in vitro, aunque en 2004 las implicaciones de la estructura in vivo seguían sin esclarecer, especialmente porque la estructura de las uniones es alterada a menudo por las proteínas que se unen a ella.[4]
Referencias[editar]
- ↑ O amontoamento pi (pi-stacking) son as interaccións non covalentes atractivas entre aneis aromáticos, e é moi importante entre os aneis das nucleobases do ADN e ARN.
- ↑ a b c d e f g h i j k l m n «Structures of helical junctions in nucleic acids». Quarterly reviews of biophysics 33 (2): 109-159. 2000. PMID 11131562. doi:10.1017/S0033583500003590.
- ↑ Nucleic acids: structures, properties, and functions. Sausalito, California: University Science Books. 2000. p. 468. ISBN 0935702490.
- ↑ a b c d e f g h Liu Y, West S (2004). «Happy Hollidays: 40th anniversary of the Holliday junction». Nat Rev Mol Cell Biol 5 (11): 937-44. PMID 15520813. doi:10.1038/nrm1502.
- ↑ Sung; Klein, H (October 2006). «Mechanism of homologous recombination: mediators and helicases take on regulatory functions». Nature Reviews Molecular Cell Biology 7 (10): 739-750. PMID 16926856. doi:10.1038/nrm2008.
- ↑ Hartel, Daniel L.; Ruvolo, Maryellen (2012). Genetics: Analysis of Genetics and Genomes. Burlington: Jones & Bartlett.
- ↑ Helleday, T. «Double-Strand Break Repair via Double Holliday Junctions (Szostak Model)». Animation. MIT.
- ↑ «Comparative and evolutionary analysis of the bacterial homologous recombination systems». PLoS Genetics 1 (2): e15. August 2005. PMC 1193525. PMID 16132081. doi:10.1371/journal.pgen.0010015.
- ↑ «43». Medical Microbiology (4th edición). University of Texas Medical Branch at Galveston. 1996. ISBN 0-9631172-1-1.
- ↑ West SC (2003). «Molecular views of recombination proteins and their control». Nat. Rev. Mol. Cell Biol. 4 (6): 435-45. PMID 12778123. doi:10.1038/nrm1127.
- ↑ Kowalczykowski SC (2000). «Initiation of genetic recombination and recombination-dependent replication». Trends Biochem. Sci. 25 (4): 156-65. PMID 10754547. doi:10.1016/S0968-0004(00)01569-3.
- ↑ Boni, MF; de Jong, MD; van Doorn, HR; Holmes, EC; Martin, Darren P. (3 de mayo de 2010). «Guidelines for identifying homologous recombination events in influenza a virus». En Martin, Darren P., ed. PLoS ONE 5 (5): e10434. PMC 2862710. PMID 20454662. doi:10.1371/journal.pone.0010434.
- ↑ «The emergence of complexity: lessons from DNA». PLoS Biology 2 (12): 2036-2038. December 2004. PMC 535573. PMID 15597116. doi:10.1371/journal.pbio.0020431.
- ↑ a b c «Nanotechnology and the double helix». Scientific American 290 (6): 64-75. June 2004. PMID 15195395. doi:10.1038/scientificamerican0604-64.
- ↑ a b c Nadrian C. Seeman (2010). «Nanomaterials Based on DNA». Annual Review of Biochemistry 79: 65-87. doi:10.1146/annurev-biochem-060308-102244.
- ↑ «DNA Origami: The Art of Folding DNA». Angewandte Chemie Int'd Ed 51 (1): 58-66. 2012. doi:10.1002/anie.201105846. Consultado el 25 de febrero de 2015.
- ↑ a b Stahl FW (1 de octubre de 1994). «The Holliday junction on its thirtieth anniversary» (PDF). Genetics 138 (2): 241-246. PMC 1206142. PMID 7828807.
- ↑ Hays FA, Watson J, Ho PS (2003). «Caution! DNA Crossing: Crystal Structures of Holliday Junctions». J Biol Chem 278 (50): 49663-49666. PMID 14563836. doi:10.1074/jbc.R300033200.
